Editorial na téma výzkumu
Úloha AAA+ proteinů při opravách a degradaci proteinů Protein Repair and Degradation
ATPasy Associated with diverse cellular Activities (AAA+) tvoří nadrodinu proteinů, které vykonávají velké množství funkcí nezbytných pro buněčnou fyziologii, včetně kontroly homeostázy proteinů, replikace DNA, rekombinace, remodelace chromatinu, zpracování ribozomální RNA, molekulárního cílení, biogeneze organel a fúze membrán (Hanson a Whiteheart, 2005; Erzberger a Berger, 2006; Snider a kol., 2008). Členové této nadrodiny jsou definováni přítomností tzv. domény AAA+, která obsahuje kanonické Walkerovy motivy A a B potřebné pro vazbu a hydrolýzu ATP (Hanson a Whiteheart, 2005). Genomy obvykle kódují přibližně deset až několik set členů rodiny AAA+ (tabulka 1; Finn et al., 2017), přičemž se předpokládá, že každý z nich je přizpůsoben specifickým funkčním nikám, které vyžadují přesné mechanismy rozpoznávání a zpracování substrátu (Hanson a Whiteheart, 2005). Nápadná adaptivní radiace proteinů AAA+, které fungují v různých prostředích, ilustruje všestrannou využitelnost domény AAA+ (Erzberger a Berger, 2006). Proteiny AAA+ obvykle tvoří hexamerické komplexy a fungují jako motory pro remodelaci jiných proteinů, DNA/RNA nebo vícesložkových komplexů (obr. 1). Mnoho chaperonů a ATP-dependentních proteáz skutečně patří nebo má podjednotky, které patří do této nadrodiny (Obrázek 1; Olivares et al., 2016).
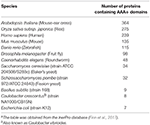
Tabulka 1. Počet proteinů AAA+ v modelových organismecha.
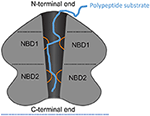
Obrázek 1. Schéma ClpA jako příklad hexameru AAA+. Schematické znázornění architektury domén a interakcí ClpA jako příkladu AAA+ hexameru se dvěma AAA+ doménami na monomer. Zde je zobrazen boční výřez hexameru ClpA, který znázorňuje centrální polypeptidový vodivý kanál. ClpA obsahuje tři domény, včetně N-koncové domény a dvou AAA+ domén: nukleotid-vazebnou doménu 1 a 2 (NBD1 a NBD2). N-koncová doména interaguje s regulátory substrátové specifity, zatímco C-konec interaguje s komorovou proteázou ClpP. Pohyblivé smyčky z NBD1 a NBD2 (oranžová) vyčnívají do centrálního kanálu a zapojují polypeptidový substrát (modrá), čímž umožňují spojení hydrolýzy ATP s translokací polypeptidu přes centrální kanál.
V posledních letech došlo k výraznému pokroku v identifikaci struktury a funkčního mechanismu velkého počtu proteinů AAA+ (Gates et al., 2017; Puchades et al., 2017; Ripstein et al., 2017; Zehr et al., 2017). V tomto výzkumném tématu je několik prvků tohoto vzrušujícího pokroku zprostředkováno v 21 článcích, které zahrnují podrobný strukturní a mechanistický pohled na několik AAA+ chaperonů a proteáz, včetně např: ClpX (Alhuwaider a Dougan; Bittner et al.; Elsholz et al.; LaBreck et al.; Vass et al.), ClpA (Bittner et al.; Duran et al.), ClpB a Hsp104 (Chang et al.; Duran et al.; Franke et al.; Johnston et al.), Hsp78 (Abrahão et al.), ClpC (Alhuwaider a Dougan; Elsholz et al.), ClpE (Elsholz et al.), Pontin (Mao a Houry), Reptin (Mao a Houry), FtsH (Alhuwaider a Dougan), 19S proteasom (Snoberger et al.; Yedidi et al.), Lon (Alhuwaider a Dougan; Bittner et al.; Fishovitz et al.), p97 (Hänzelmann a Schindelin; Saffert et al.; Ye et al.), Pex1/6 (Saffert et al.), CbbQ (Mueller-Cajar), rubisco aktiváza (Bhat et al.), torsiny (Chase et al.) a mitochondriální AAA+ proteázy (Glynn). Zde představujeme tyto fascinující práce.
Studie o ClpXP, Lon a příbuzných proteázách závislých na ATP
Ve svém výzkumném článku „The Protein Chaperone ClpX Targets Native and Non-native Aggregated Substrates for Remodeling, Disassembly, and Degradation with ClpP,“ LaBreck et al. provádějí řadu elegantních experimentů, aby zjistili, že ClpX má disagregázovou aktivitu vůči polypeptidům, které obsahují specifické signály rozpoznávání ClpX (LaBreck et al.). V přítomnosti ClpP ClpX spojuje disagregaci těchto substrátů s jejich degradací. Důležité je také zjištění, že ClpXP zabraňuje hromadění agregátů tvořených proteiny nesoucími rozpoznávací signály ClpX in vivo (LaBreck et al.). Tyto studie osvětlují ClpX jako proteinovou disagregázu, která byla dříve podceňována.
V článku „The Essential Role of ClpXP in Caulobacter crescentus Requires Species Constrained Substrate Specificity“ zkoumají Vass a kol. druhově specifické funkce ClpX (Vass a kol.). Je zajímavé, že ClpX je nezbytný u některých druhů, jako je C. crescentus, ale není nezbytný u jiných bakterií, jako je E. coli (Vass et al.). Důležité je, že ClpX E. coli nebyl schopen doplňovat ClpX C. crescentus in vivo (Vass et al.). Tato nedostatečná aktivita byla způsobena druhově specifickými rozdíly v N-koncové doméně ClpX, které jsou kritické pro zpracování podjednotky DnaX zavaděče replikačních svorek u C. crescentus. Malé rozdíly ve specifičnosti ClpX tak mohou být kritické zejména pro specifické bakteriální druhy.
V přehledu „Functional Diversity of AAA+ Protease Complexes in Bacillus subtilis“ se Elsholz a kol. zabývají funkcemi několika AAA+ proteáz u B. subtilis, konkrétně: ClpCP, ClpEP, ClpXP, ClpYQ, LonA/B a FtsH (Elsholz et al.). Pojednávají o tom, jak různé stresové reakce řídí jejich expresi, a o fenotypech pozorovaných při deleci těchto různých proteáz. Popisují schopnost některých z těchto proteáz řídit kompetici, sporulaci, motilitu a tvorbu biofilmu. Nakonec autoři diskutují o cílení těchto proteáz pro vývoj nových antibiotik.
V přehledu nazvaném „AAA+ Machines of Protein Destruction in Mycobacteria“ se Alhuwaider et al. (Alhuwaider a Dougan) zabývají nedávným pokrokem při určování struktury a funkce AAA+ proteáz mykobakterií. Těmito proteázami jsou: ClpXP1P2, ClpC1P1P2, Lon, FtsH a Mpa. Autoři se rovněž zabývají systémem Pup-proteasom (PPS) přítomným u mykobakterií, který je ekvivalentem ubikvitin-proteasomového systému u eukaryot. Alhuwaider a kol. pak v závěru diskutují o nových sloučeninách, které dysregulují nebo inhibují aktivitu ClpP1P2, a dalších, které dysregulují ClpC1. Tyto sloučeniny mají slibnou aktivitu proti mykobakteriím.
Ve výzkumném článku s názvem „The Copper Efflux Regulator CueR Is Subject to ATP-Dependent Proteolysis in Escherichia coli“ Bittner et al. dokazují, že AAA+ proteázy Lon, ClpXP a ClpAP jsou zodpovědné za degradaci E. coli CueR, což je transkripční faktor, který řídí indukci systému Cue efluxu mědi (Bittner et al.). Autoři zjistili, že rozpoznání CueR proteázami AAA+ vyžaduje přístupný C-konec CueR. Došli k závěru, že pro homeostázu mědi v E. coli jsou nutné ATP-dependentní proteázy.
Fishovitz a kol. provádějí podrobné srovnání mezi lidskou a E. coli Lon ve svém výzkumném článku s názvem „Využití mechanistické enzymologie k hodnocení významu vazby ADP na lidskou Lon proteázu“ (Fishovitz a kol.). Pomocí podrobné mechanistické studie zjistili, že na rozdíl od E. coli Lon má lidská Lon nízkou afinitu k ADP, přestože vykazuje srovnatelné hodnoty kcat a KM v ATPázové aktivitě. Navrhují, že lidský Lon není regulován mechanismem výměny ADP/ATP podporovaným substrátem. Tyto rozdíly mezi lidskou a E. coli Lon by mohly v budoucnu umožnit vývoj druhově specifických inhibitorů Lon.
Dr. Glynn se ve svém přehledu „Multifunctional Mitochondrial AAA Proteases“ zabývá dvěma mitochondriálními proteázami AAA, i-AAA a m-AAA (Glynn). Obě jsou proteiny vnitřní membrány mitochondrií. Proteáza i-AAA však promítá domény ATPázy a proteázy do mitochondriálního mezimembránového prostoru, zatímco proteáza m-AAA promítá katalytické domény do matrix. Jsou diskutovány struktury těchto proteáz a mechanismus jejich funkce. Proteázy mohou provádět kompletní degradaci substrátu, ale také mohou štěpit pouze určité substráty, jako například u MrpL32 a Atg32.
ClpB a Hsp104
V minirecenzi „Structural Elements Regulating AAA+ Protein Quality Control Machines“ (Strukturní prvky regulující stroje pro kontrolu kvality proteinů AAA) Chang et al. pojednávají o tom, jak mohou pórová smyčka-1, mezipodjednotkový signální motiv a vložený motiv Pre-Sensor I přispívat k aktivitě dvou disagregáz Hsp100, bakteriální ClpB a kvasinkové Hsp104 (Chang et al.). Navrhují model, jak by tyto strukturní prvky mohly umožnit spojení cyklu AAA+ ATPázy s translokací substrátu přes centrální kanál ClpB a Hsp104. Předpokládá se, že tento proces translokace polypeptidů je základem toho, jak ClpB a Hsp104 extrahují polypeptidy z agregovaných struktur (Chang et al.).
Duran et al. ve svém přehledu uvádějí „Comparative Analysis of the Structure and Function of AAA+ Motors ClpA, ClpB, and Hsp104: Common Threads and Disparate Functions“ (Duran et al.). Pojednávají o schopnosti těchto tří proteinů AAA+ (ClpA, ClpB a Hsp104) přemisťovat polypeptidy prostřednictvím svých hexamerických komplexů. Všechny tyto proteiny mají dvě domény AAA+ a je o nich známo, že rozkládají proteiny. Důležité je, že ClpB a Hsp104 fungují také jako disagregázy, zatímco ClpA může tvořit komplex s proteázou ClpP. Autoři zdůrazňují potřebu použít kinetické metody přechodného stavu ke zkoumání kinetických mechanismů těchto motorických proteinů. Popisují, jak jim použití těchto metod umožnilo ukázat, že například ClpA translokuje polypeptidy rychlostí přibližně 20 aa s-1, zatímco v komplexu s proteázou ClpP je rychlost translokace ClpA ještě vyšší, přibližně 35 aa s-1. Autoři se také zabývají významem chaperonu Hsp70 pro funkci ClpB/Hsp104 a pozorováním druhové specifičnosti v interakci mezi Hsp70 a ClpB/Hsp104.
V článku s názvem „Mutant Analysis Reveals Allosteric Regulation of ClpB Disaggregase“ provádějí Franke a kol. mutační analýzu disagregasy E. coli ClpB, aby charakterizovali její alosterickou regulaci (Franke et al.). ClpB lze rozdělit na N-koncovou doménu a dvě AAA+ domény oddělené šroubovicovou oblastí označovanou jako M-doména. Autoři identifikovali vysoce konzervovaný zbytek v první doméně AAA+, A328. Bylo zjištěno, že mutant ClpB-A328V má velmi vysokou ATPázovou aktivitu a vykazuje buněčnou toxicitu. Neočekávaně byla vysoká ATPázová aktivita ClpB-A328V způsobena především druhým AAA+ kruhem, jak bylo zjištěno pomocí hmotnostní spektrometrie s amidovou vodíkovou výměnou. Autoři dospěli k závěru, že A328 je klíčovým zbytkem při řízení hydrolýzy ATP v obou AAA+ kruzích ClpB.
Ve svém výzkumném článku s názvem „Substrate Discrimination by ClpB and Hsp104“ popisují Johnston a kol. vrozené substrátové preference ClpB a Hsp104 v nepřítomnosti chaperonových systémů DnaK a Hsp70 (Johnston a kol.). Ukazují, že substrátová specifita je určena první doménou AAA+ v každém proteinu. K tomuto závěru dospěli testováním schopnosti obou chaperonů působit na několik modelových substrátů. Testovali také různé chiméry obou chaperonů.
V článku „Hsp78 (78 kDa Heat Shock Protein), a representative AAA Family Member Found in the Mitochondrial Matrix of Saccharomyces cerevisiae“ se Abrahão et al. zabývají strukturou a funkcí Hsp78 (Abrahao et al.). Hsp78 je mitochondriální paralog Hsp104, který funguje při disagregaci a reaktivaci proteinů (Abrahao et al.). Zajímavé je, že Hsp104 a Hsp78 byly ztraceny při přechodu od prvoků k metazoím (Abrahao et al.). Abrahao et al. však hovoří o existenci ANKCLP, který se objevuje vedle Hsp78 a Hsp104 u prvoků a přežil evoluční přechod k metazoím. ANKCLP má doménu AAA+ podobnou nukleotid-vázající doméně 2 (NBD2) Hsp104 a Hsp78, ale jinak se značně liší. Zajímavé je, že mutace v ANKCLP způsobují u lidí 3-methylglutakonovou acidurii, progresivní atrofii mozku, intelektuální postižení, vrozenou neutropenii, kataraktu a pohybové poruchy (Abrahao et al.).
p97
Saffert et al. ve svém přehledu „Structure and Function of p97 and Pex1/6 Type II AAA+ Complexes“ pojednávají o dvou různých AAA+ komplexech, které remodelují ubikvitinované substrátové proteiny (Saffert et al.). Jednou z funkcí p97 je dislokace ubikvitinovaných substrátů z membrány ER do proteazomu během degradace spojené s ER (Saffert et al.). Naproti tomu Pex1/Pex6 je heterohexamerický motor složený ze střídajících se podjednotek Pex1 a Pex6, který je nezbytný pro biogenezi a funkci peroxizomů. Jsou diskutovány nejnovější struktury p97 a Pex1/6 získané kryoelektronovou mikroskopií (kryoEM) a zdůrazněny klíčové strukturní rozdíly.
V přehledu nazvaném „A Mighty ‚Protein Extractor‘ of the Cell: Structure and Function of the p97/CDC48 ATPase“ shrnují Ye et al. současné poznatky o struktuře a funkci p97 a její roli u několika chorob (Ye et al.). p97 má dvě domény AAA+ spojené krátkým linkerem. Má také N-koncovou doménu, která zprostředkovává její interakce s různými adaptorovými proteiny. Autoři podrobně rozebírají strukturu p97 a vliv nukleotidů na jeho různé konformace. Tyto studie jsou založeny na použití technik, jako je EM, rentgenová krystalografie a vysokorychlostní mikroskopie atomárních sil. Autoři poté diskutují o mnohobuněčných funkcích tohoto vysoce konzervovaného proteinu, včetně jeho role v degradaci proteinů asociované s ER (ERAD), degradaci asociované s mitochondriemi (MAD) extrakcí polypeptidů z vnější mitochondriální membrány a degradaci asociované s ribozomy (RAD). Nakonec Ye et al. uvádí přehled mutací p97 vedoucích k několika lidským onemocněním, jako je IBMPFD (Inclusion Body Myopathy associated with Paget’s disease of the bone and Frontotemporal Dementia)], FALS (familiární amyotrofická laterální skleróza), CMT2Y (Charcot-Marie-Tooth disease, typ 2Y), hereditární spastické paraplegie (HSP), Parkinsonova choroba (PD) a Alzheimerova choroba (AD).
Hanzelmann a Schindelin ve svém přehledu o p97 nazvaném „The Interplay of Cofactor Interactions and Post-translational Modifications in the Regulation of the AAA+ ATPase p97“ pojednávají o tom, jak různé kofaktory modulují aktivitu ATPázy p97 (Hanzelmann a Schindelin). Zdůrazňují skutečnost, že schopnost p97 podílet se na velkém množství buněčných procesů je dána velkým počtem kofaktorů, které s tímto proteinem interagují. Objasnili tři různé třídy kofaktorů p97, a to: (i) kofaktory získávající substrát, jako jsou proteiny UBA-UBX a UFD1-NPL4, (ii) kofaktory zpracovávající substrát, jako jsou ubikvitinové (E3) ligázy a deubikvitinázy (DUB), a (iii) regulační kofaktory, jako jsou proteiny UBX, které mohou sekvestrovat nebo recyklovat hexamery p97. Autoři se rovněž zabývají úlohou posttranslačních modifikací na aktivitu p97 a na jeho interakce s kofaktory a substráty.
AAA+ proteiny proteazomu
V článku „AAA-ATPases in Protein Degradation“ (AAA-ATPázy v degradaci proteinů) se Yedidi et al. podávají přehled aktivit Rpt1, Rpt2, Rpt3, Rpt4, Rpt5 a Rpt6, což jsou AAA+-ATPázy eukaryotického proteazomu, a také některých jejich bakteriálních příbuzných, jako jsou PAN, Mpa a VAT (Yedidi et al.). Zaměřují se na nové technologie, aby pochopili, jak tyto AAA+ ATPázy fungují tím, že přemisťují nesložené polypeptidy do proteolytické komory proteázy (Yedidi et al.). Zdá se, že konformační změny v kruhu AAA+ a přilehlé komůrce proteázy vytvářejí peristaltický čerpací mechanismus, který dopravuje substráty k degradaci (Yedidi et al.).
Ve svém výzkumném článku „The Proteasomal ATPases Use a Slow but Highly Processive Strategy to Unfold Proteins“ (Proteazomální ATPázy používají pomalou, ale vysoce procesní strategii k rozbalování proteinů) Snoberger et al. zjistili, že proteazomální proteiny AAA+ využívají k dopravě substrátů do proteolytické dutiny proteazomu nízkorychlostní, ale vysoce procesní motorický mechanismus (Snoberger et al.). Tento mechanismus je v kontrastu s mechanismem ClpX, který využívá vysokorychlostní, ale méně procesní motorický mechanismus k dopravě substrátů k proteáze ClpP k degradaci. Tyto rozdíly v motorickém mechanismu se mohly vyvinout v reakci na rozdílné požadavky jejich specifické klientely.
Rubisco aktivázy
V přehledu na téma „Rubisco aktivázy: Bhat et al. se zabývají jedinečnou funkcí Rubisco aktivázy (Rca) při remodelaci Rubisco (Bhat et al.). Rca je chaperon AAA+, který je vysoce konzervovaný u fotosyntetických organismů od bakterií po vyšší rostliny. Rubisco je enzym ribulosa-1,5-bisfosfátkarboxyláza/oxygenáza, který se podílí na fixaci atmosférického CO2 během fotosyntézy. Je to nejrozšířenější protein na Zemi a je klíčovým enzymem při syntéze všech organických látek na planetě. Rubisco je však chudý enzym a je snadno inhibován vedlejšími produkty svých katalytických reakcí nebo sloučeninami syntetizovanými některými rostlinami za nízké hladiny osvětlení. Rca funguje tak, že zmírňuje nebo „léčí“ Rubisco z těchto problematických inhibic. Autoři se zabývají strukturou Rca z různých druhů a možnými mechanismy jeho funkce.
Dr. Mueller-Cajar uvádí přehled „The Diverse AAA+ Machines that Repair Inhibited Rubisco Active Sites“ (Mueller-Cajar). Pojednává o přítomnosti tří evolučně odlišných tříd aktiváz Rubisco (Rcas): (1) Rcas zeleného a (2) červeného typu, které se většinou vyskytují u fotosyntetických eukaryot zelené a červené plastidové linie, a (3) CbbQO přítomné u chemoautotrofních bakterií. Pojednává o evoluci těchto aktiváz a jejich možném využití v syntetické biologii ke zvýšení aktivity Rubisco v rostlinách.
Torsin
V perspektivním článku „Torsin ATPases: Chase a kol. pojednávají o Torsinech, které jsou také fylogeneticky příbuzné s NBD2 kvasinkového Hsp104 (Chase et al.). Torsiny jsou jediné AAA+ ATPázy lokalizované uvnitř ER a spojeného jaderného obalu (Chase et al.). Zajímavé je, že mutace v TorsinA způsobují dystonii DYT1, neurologickou poruchu u lidí (Chase et al.). Torsiny vykazují slabou ATPázovou aktivitu, která je zvýšena prostřednictvím komplementace aktivního místa díky společnému sdružování se specifickými akcesorními kofaktory LAP1 a LULL1 (Chase et al.). Chase et al. naznačují, že dynamické sestavování a rozkládání komplexů Torsin/kofaktor hraje důležitou roli v jejich funkci při jaderném obchodu a sestavování komplexů jaderných pórů (Chase et al.).
Pontin a Reptin
Mao a Houry ve svém rozsáhlém přehledu na téma „Úloha Pontinu a Reptinu v buněčné fyziologii a etiologii rakoviny“ pojednávají o mnohostranných funkcích vysoce konzervovaných AAA+ ATPáz Pontinu a Reptinu (Mao a Houry). Tyto dva proteiny obvykle fungují společně jako komplex, ale mohou fungovat i samostatně. Autoři zdůrazňují roli Pontinu a Reptinu v remodelaci chromatinu. Pojednávají také o tom, jak Pontin a Reptin modulují transkripční aktivity několika protoonkogenů, jako jsou MYC a β-katenin. Mao a Houry objasňují, jak bylo zjištěno, že Pontin a Reptin jsou nezbytné pro sestavení signálních komplexů PIKK a také telomerázy, mitotického vřeténka, RNA polymerázy II a snoRNPs. V závěru autoři uvádějí přehled současných snah zaměřených na identifikaci inhibitorů Pontinu a Reptinu, které mají být vyvinuty jako nové protinádorové látky.
Závěrečné poznámky
Na závěr tento soubor 21 článků poukazuje na řadu důležitých strukturních a mechanistických aspektů proteinů AAA+, které se podílejí na opravách a degradaci proteinů. S napětím očekáváme, jak se bude tento obor dále rozvíjet během probíhající kryoEM revoluce (Egelman, 2016). Očekáváme, že kryo-EM umožní hlubší pochopení toho, jak tyto fascinující molekulární stroje fungují v různých situacích (Gates et al., 2017; Puchades et al., 2017; Ripstein et al., 2017; Zehr et al., 2017).
Příspěvky autorů
Všichni uvedení autoři se na práci významně, přímo a intelektuálně podíleli a schválili ji k publikaci.
Prohlášení o střetu zájmů
Autoři prohlašují, že výzkum byl prováděn bez jakýchkoli komerčních nebo finančních vztahů, které by mohly být chápány jako potenciální střet zájmů.
Poděkování
Práce v laboratoři WH v této oblasti je financována z projektového grantu Canadian Institutes of Health Research (PJT-148564). JS je podporován grantem NIH R01GM099836.
Egelman, E. H. (2016). Současná revoluce v kryoEM. Biophys. J. 110, 1008-1012. doi: 10.1016/j.bpj.2016.02.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Erzberger, J. P., a Berger, J. M. (2006). Evoluční vztahy a strukturní mechanismy proteinů AAA+. Annu. Rev. Biophys. Biomol. Struct. 35, 93-114. doi: 10.1146/annurev.biophys.35.040405.101933
PubMed Abstract | CrossRef Full Text | Google Scholar
Finn, R. D., Attwood, T. K., Babbitt, P. C., Bateman, A., Bork, P., Bridge, A. J., et al. (2017). InterPro v roce 2017 – nad rámec anotací proteinových rodin a domén. Nucleic Acids Res. 45, D190-D199. doi: 10.1093/nar/gkw1107
PubMed Abstract | CrossRef Full Text | Google Scholar
Gates, S. N., Yokom, A. L., Lin, J., Jackrel, M. E., Rizo, A. N., Kendsersky, N. M., et al. (2017). Ratchet-like polypeptidový translokační mechanismus AAA+ disagregázy Hsp104. Science 357, 273-279. doi: 10.1126/science.aan1052
PubMed Abstract | CrossRef Full Text | Google Scholar
Hanson, P. I., and Whiteheart, S. W. (2005). Proteiny AAA+: mít motor, bude fungovat. Nat. Rev. Mol. Cell Biol. 6, 519-529. doi: 10.1038/nrm1684
PubMed Abstract | CrossRef Full Text | Google Scholar
Olivares, A. O., Baker, T. A., and Sauer, R. T. (2016). Mechanistické poznatky o bakteriálních AAA+ proteázách a strojích na přetváření proteinů. Nat. Rev. Microbiol. 14, 33-44. doi: 10.1038/nrmicro.2015.4
PubMed Abstract | CrossRef Full Text | Google Scholar
Puchades, C., Rampello, A. J., Shin, M., Giuliano, C. J., Wiseman, R. L., Glynn, S. E., et al. (2017). Struktura mitochondriální vnitřní membrány AAA+ proteázy YME1 umožňuje nahlédnout do zpracování substrátu. Science 358:eaao0464. doi: 10.1126/science.aao0464
PubMed Abstract | CrossRef Full Text | Google Scholar
Ripstein, Z. A., Huang, R., Augustyniak, R., Kay, L. E., and Rubinstein, J. L. (2017). Struktura AAA+ unfoldázy v procesu rozkládání substrátu. Elife 6:e25754. doi: 10.7554/eLife.25754
PubMed Abstract | CrossRef Full Text | Google Scholar
Snider, J., Thibault, G., and Houry, W. A. (2008). Nadrodina funkčně rozmanitých proteinů AAA+. Genome Biol. 9:216. doi: 10.1186/gb-2008-9-4-216
PubMed Abstract | CrossRef Full Text | Google Scholar
Zehr, E., Szyk, A., Piszczek, G., Szczesna, E., Zuo, X., and Roll-Mecak, A. (2017). Kataninová spirální a prstencová struktura vrhá světlo na výkonový tah pro oddělování mikrotubulů. Nat. Struct. Mol. Biol. 24, 717-725. doi: 10.1038/nsmb.3448
PubMed Abstract | CrossRef Full Text | Google Scholar
.