Jednonukleotidový polymorfismus
Jednonukleotidový polymorfismus (SNP) označuje rozdíl jednoho nukleotidu na stejné pozici v sekvenci genomové DNA. Obecně se odchylka jednoho nukleotidu s frekvencí vyšší než 1 % nazývá SNP. SNP zahrnují pouze odchylku jedné báze, která může být způsobena přechodem jedné báze nebo transverzí. V lidském genomu je přibližně jeden SNP na 1000 bází a celkový počet SNP v lidském genomu je přibližně 3 x 106.
SNP se stal třetí generací genetických markerů. Díky svým vlastnostem jsou SNP vhodnější pro studium genetické pleiotropie u komplexních znaků a nemocí a pro rozpoznávání genů v populaci než jiné polymorfní markery. SNP mají značný význam v biomedicínském výzkumu. SNP byly použity v celogenomových asociačních studiích jako markery s vysokým rozlišením při mapování genů souvisejících s nemocemi nebo normálními znaky. SNP bez pozorovatelného vlivu na fenotyp (tzv. tiché mutace) mohou být rovněž užitečné díky svému množství a stabilnímu dědění v průběhu generací.
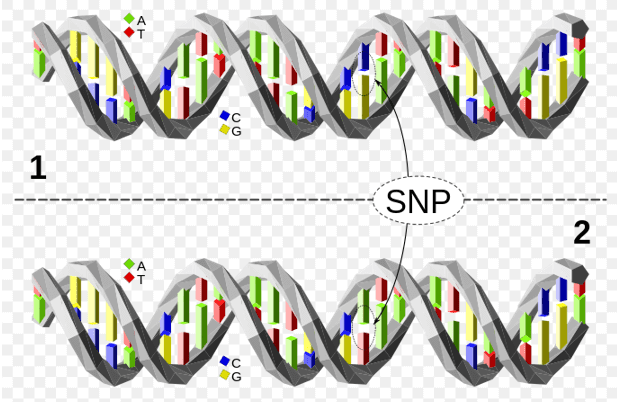 Obrázek 1. SNP mikročipy SNP. Horní molekula DNA se liší od dolní molekuly DNA v místě jednoho páru bází (polymorfismus C/A).
Obrázek 1. SNP mikročipy SNP. Horní molekula DNA se liší od dolní molekuly DNA v místě jednoho páru bází (polymorfismus C/A).
Mikroarray
Mikroarray DNA označuje genový čip s velkým počtem sondových sekvencí DNA ve specifickém uspořádání imobilizovaných na pevném substrátu. Principem je teorie hybridizace nukleových kyselin a detekovaná DNA vzorku je hybridizována s DNA microarray a prodloužena. Následně se fragmenty nekomplementární vazebné reakce na čipu smyjí a genový čip se podrobí laserovému konfokálnímu skenování. Intenzita fluorescenčního signálu je poté změřena a interpretována jako abundance různých genů prostřednictvím určitého zpracování dat.
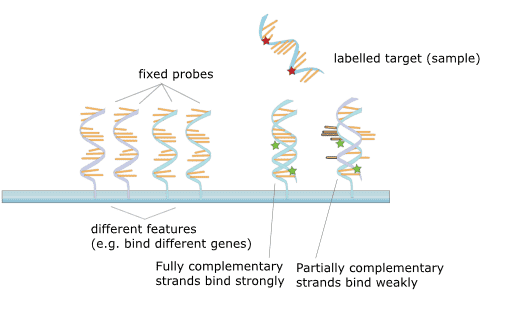 Obrázek 2. Princip mikroarray.
Obrázek 2. Princip mikroarray.
Mikroarray SNP
Mikroarray SNP používá známé sekvence nukleotidů jako sondy, které hybridizují s testovanými sekvencemi DNA, což umožňuje kvalitativní a kvantitativní analýzu SNP prostřednictvím detekce signálu. Ve srovnání s tradiční metodou diagnostiky jednotlivých buněk je SNP microarray vysoce výkonnou metodou, která je schopna provést tisíce reakcí na povrchu oligonukleotidového čipu najednou.
Pracovní postup SNP microarray
Obecný pracovní postup SNP microarray je znázorněn na obrázku 3.
. Stručně řečeno, existuje několik procesů: Výroba čipů SNP, příprava genomové DNA vzorku, hybridizace a fluorescenční skenování.
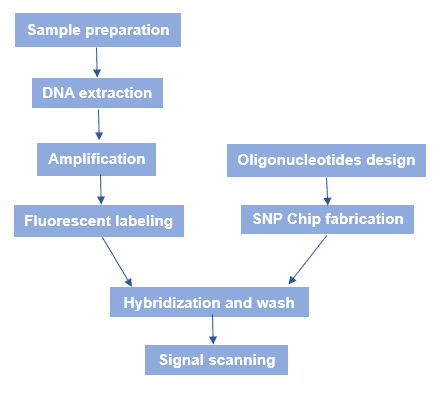 Obrázek 3. Pracovní postup mikročipů SNP.
Obrázek 3. Pracovní postup mikročipů SNP.
- Výroba čipů SNP
Předem navržené čipy oligonukleotidů nebo cDNA jsou uspořádány uspořádaně a ve vysoké hustotě na skleněném nosiči, aby se vytvořily mikročipy nebo jádrové čipy. Metody výroby čipu zahrnují světlem řízenou syntézu in-situ, metodu chemického nástřiku, metodu kontaktního bodového povlakování, syntézu řízenou DNA in-situ, metodu bezkontaktního mikromechanického tisku TOPSPOT® a reprodukci měkkou litografií atd. Na čip o ploše 1 cm2 lze nyní umístit celkem 4×105 různých molekul DNA. Technologie Affymetrix GeneChip® umožňuje syntézu oligonukleotidů s vysokou hustotou in situ a je v této oblasti průkopníkem. - Příprava genomické DNA vzorku
Nejprve příprava materiálu vzorku. Poté se genomová DNA extrahuje a amplifikuje pomocí PCR. Nakonec se k jejímu značení použijí různá fluorescenční barviva. - Hybridizace a skenování
Fluorescenčně značená genomová DNA se za vhodných reakčních podmínek hybridizuje s mikročipy SNP. Po promytí se ke skenování fluorescence použije specifický fluorescenční skener. Poté se provede počítačová analýza obrazu pro zpracování dat před provedením bioinformatické analýzy cílového genu.
V CD Genomics se věnujeme poskytování spolehlivých služeb genotypování SNP, včetně genotypování pomocí sekvenování, SNP microarray, MassARRAY SNP genotypování, Hi-SNPseq, SNaPshot Multiplex System pro SNP genotypování a TaqMan SNP genotypování.
- Iwamoto K, Bundo M, Ueda J, et al. Detekce chromozomálních strukturálních změn v jednotlivých buňkách pomocí SNP array: systematický průzkum amplifikačního zkreslení a optimalizace pracovního postupu. Plos One, 2007, 2(12): e1306.
- Ho C C, Mun K S, Naidu R. SNP array technology: an array of hope in breast cancer research. Malaysian Journal of Pathology, 2013, 35(1):33-43.
- Ahmad A, Iqbal M A. Significance of genome-wide analysis of copy number alterations and UPD in myelodysplastic syndromes using combined CGH-SNP arrays. Current Medicinal Chemistry, 2012, 19(22).
- Sund K L, Zimmerman S L, Thomas C, et al. Regions of homozygosity identified by SNP microarray analysis aid in the diagnosis of autosomal recessive disease and incidentally detect parental blood relationships. Genetics in Medicine Official Journal of the American College of Medical Genetics, 2013, 15(1):70-78.
- Liu S, Zhou Z, Lu J, et al. Generation of genome-scale gene-associated SNPs in catfish for the construction of a high-density SNP array. Bmc Genomics, 2011, 12(1):53.