Single nucleotide polymorphism
Single nucleotide polymorphism (SNP) henviser til forskellen på et enkelt nukleotid på samme position i den genomiske DNA-sekvens. Generelt kaldes en enkelt nukleotidvariation med en frekvens på mere end 1 % for en SNP. SNP’er omfatter kun en enkelt basevariation, som kan være forårsaget af en enkelt baseovergang eller transversion. Der er ca. én SNP pr. 1000 baser i det menneskelige genom, og det samlede antal SNP’er i det menneskelige genom er ca. 3 x 106.
SNP er blevet den tredje generation af genetiske markører. SNP’ernes egenskaber gør dem mere velegnede til undersøgelser af genetisk pleiotropi i komplekse træk og sygdomme og populationsbaseret gengenkendelse end andre polymorfe markører. SNP’er har stor betydning for den biomedicinske forskning. SNP’er er blevet anvendt i genomdækkende associationsundersøgelser som højopløselige markører i genkortlægning i forbindelse med sygdomme eller normale egenskaber. SNP’er uden en observerbar indvirkning på fænotypen (kaldet tavse mutationer) kan også være nyttige på grund af deres mængde og stabile nedarvning over generationer.
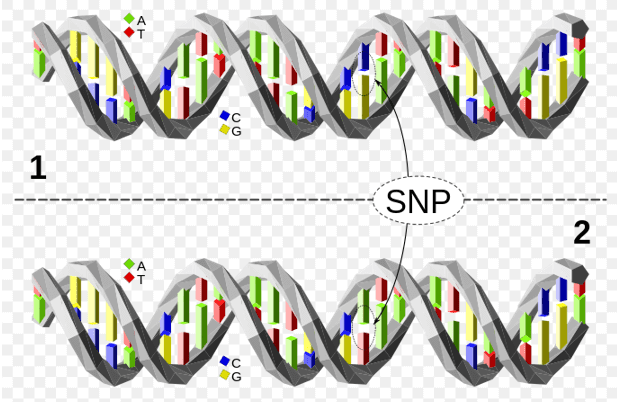 Figur 1. Det øverste DNA-molekyle adskiller sig fra det nederste DNA-molekyle på et enkelt basepars placering (en C/A-polymorfi).
Figur 1. Det øverste DNA-molekyle adskiller sig fra det nederste DNA-molekyle på et enkelt basepars placering (en C/A-polymorfi).
Mikroarray
DNA-mikroarrayet henviser til en genchip med et stort antal probe-DNA-sekvenser i en specifik opstilling immobiliseret på et fast substrat. Princippet er nukleinsyrehybridiseringsteorien, og det detekterede prøve-DNA hybridiseres med DNA-mikroarrayet og forlænges. Efterfølgende vaskes fragmenterne fra den ikke-komplementære bindingsreaktion på chippen væk, og genchippen underkastes konfokal laserscanning. Fluorescenssignalintensiteterne måles derefter og fortolkes som hyppigheden af forskellige gener gennem en vis databehandling.
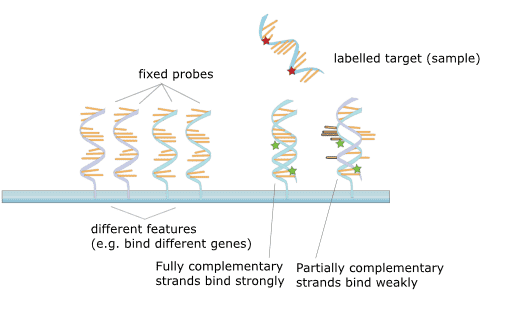 Figur 2. Princippet for mikroarray.
Figur 2. Princippet for mikroarray.
SNP-mikroarray
SNP-mikroarray anvender kendte nukleotidsekvenser som prober, der hybridiseres med de testede DNA-sekvenser, hvilket giver mulighed for kvalitativ og kvantitativ SNP-analyse gennem signaldetektion. Sammenlignet med den traditionelle enkeltcellediagnostiske metode er SNP-mikroarray en metode med højt gennemløb, som er i stand til at udføre tusindvis af reaktioner på overfladen af oligonukleotidchippen på én gang.
Arbejdsgangen for SNP-mikroarray
Den generelle arbejdsgang for SNP-mikroarray er vist i figur 3. Kort fortalt er der flere processer: SNP-chipfremstilling, forberedelse af genomisk DNA, hybridisering og fluorescensscanning.
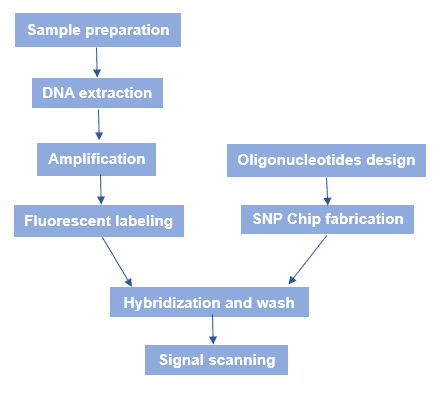 Figur 3. Arbejdsgangen for SNP-mikroarray.
Figur 3. Arbejdsgangen for SNP-mikroarray.
- Fremstilling af SNP-chips
De på forhånd designede oligonukleotid- eller cDNA-chips arrangeres ordnet og i høj tæthed på glasbæreren for at fremstille mikroarrays eller kernechips. Fremstillingsmetoderne for chippen omfatter lysstyret in-situ-syntese, kemisk spraymetode, kontaktpunktbelægningsmetode, in-situ DNA-kontrolleret syntese, kontaktløs mikromekanisk trykmetode TOPSPOT® og blød litografireproduktion osv. Der kan nu anbringes i alt 4 x 105 forskellige DNA-molekyler på en chip på 1 cm2. Affymetrix GeneChip®-teknologien muliggør in situ-oligonukleotidsyntese med høj tæthed og har været en pioner på dette område. - Prøvepræparering af genomisk DNA
Først skal prøvematerialet forberedes. Derefter ekstraheres det genomiske DNA og forstærkes ved PCR. Endelig anvendes forskellige fluorescerende farvestoffer til at markere det. - Hybridisering og scanning
Det genomiske DNA, der er mærket med fluorescens, hybridiseres med SNP-mikroarray under passende reaktionsbetingelser. Efter at være blevet vasket, anvendes en specifik fluorescensscanner til at scanne fluorescensen. Derefter foretages computerbilledanalysen for at behandle dataene, inden målgenet bioinformatisk analyse udføres.
På CD Genomics er vi dedikeret til at levere pålidelige SNP-genotyperingstjenester, herunder genotypning ved sekventering, SNP-mikroarray, MassARRAY SNP-genotypning, Hi-SNPseq, SNaPshot Multiplex System til SNP-genotypning og TaqMan SNP-genotypning.
- Iwamoto K, Bundo M, Ueda J, et al. Påvisning af kromosomale strukturelle ændringer i enkeltceller ved hjælp af SNP-arrays: en systematisk undersøgelse af amplifikationsbias og optimeret arbejdsgang. Plos One, 2007, 2(12): e1306.
- Ho C C C, Mun K S, Naidu R. SNP-array-teknologi: et håb inden for forskning i brystkræft. Malaysian Journal of Pathology, 2013, 35(1):33-43.
- Ahmad A, Iqbal M A. Significance of genome-wide analysis of copy number alterations and UPD in myelodysplastic syndromes using combined CGH-SNP arrays. Current Medicinal Chemistry, 2012, 19(22).
- Sund K L, Zimmerman S L, Thomas C, et al. Regioner af homozygotitet identificeret ved SNP-mikroarray-analyse hjælper med at diagnosticere autosomal recessiv sygdom og tilfældigvis påvise forældrenes blodforbindelser. Genetics in Medicine Official Journal of the American College of Medical Genetics, 2013, 15(1):70-78.
- Liu S, Zhou Z, Lu J, et al. Generering af gen-associerede SNP’er på genomskala i havkat til konstruktion af et SNP-array med høj tæthed. Bmc Genomics, 2011, 12(1):53.