Angesichts der relativen strukturellen Einfachheit und Wiederholbarkeit der DNA ist es naheliegend, dass Proteine, die spezifisch an sie binden, gemeinsame DNA-Bindungsdomänenmotive haben, aber mit spezifischen Aminosäureseitenketten, die spezifische Bindungsinteraktionen ermöglichen.
- Helix-Turn-Helix: kommt in prokaryotischen DNA-Bindungsproteinen vor.
Abbildung: Helix-Turn-Helix
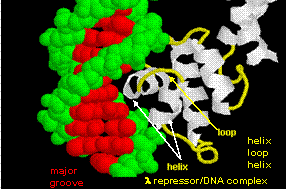
Die Abbildungen zeigen zwei solcher Proteine, den Cro-Repressor von Bakteriophage 434 und den Lambda-Repressor von Bakteriophage Lambda. (Bakteriophagen sind Viren, die Bazillen infizieren.) Beachten Sie, wie die Spezifität zum Teil durch die Bildung spezifischer H-Bindungen zwischen dem Protein und der Hauptfurche der Operator-DNA erreicht wird.
Abbildung: Lambda-Repressor/DNA-Komplex
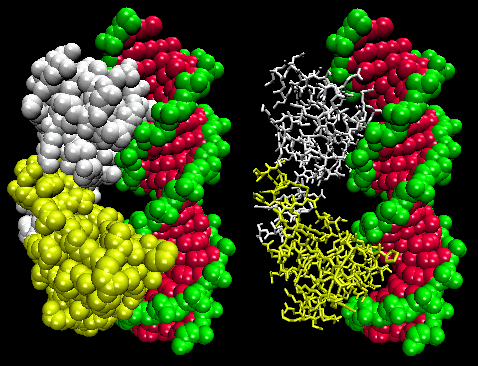
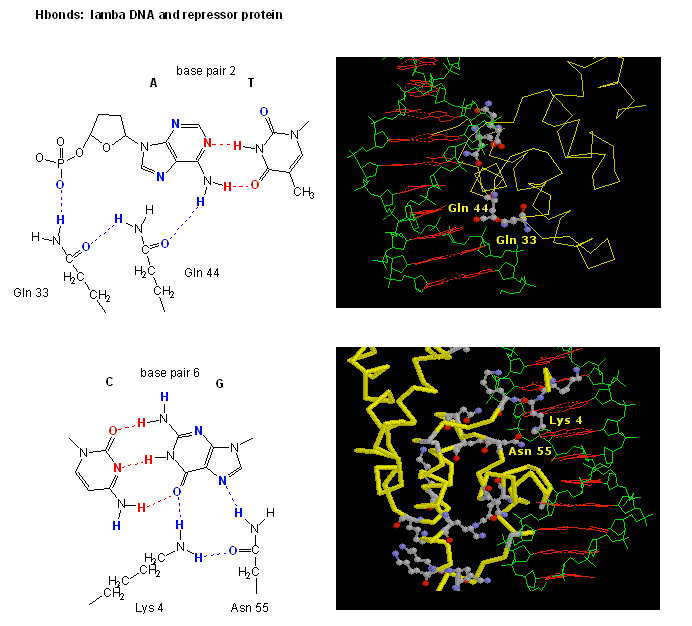
Abbildung: H-Bindung-Wechselwirkungen zwischenλ-Repressor und DNA
![]() Jmol: Aktualisierter Lambda-Repressor/DNA-Komplex Jmol14 (Java) | JSMol (HTML5)
Jmol: Aktualisierter Lambda-Repressor/DNA-Komplex Jmol14 (Java) | JSMol (HTML5)
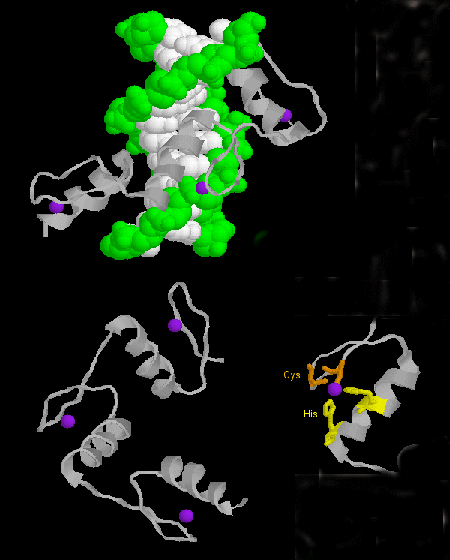
- Zinkfinger: (Eukaryoten) Diese Proteine haben ein gemeinsames Sequenzmotiv von X3-Cys-X2-4-Cys-X12-His-X3-4-His-X4-, wobei X eine beliebige Aminosäure ist. Zn2+ ist tetraedrisch mit den Cys- und His-Seitenketten koordiniert, die sich auf einem von zwei antiparallelen Betasträngen bzw. einer Alpha-Helix befinden. Der Zinkfinger, der durch das Zink stabilisiert wird, bindet an die Hauptfurche der DNA. ]
Abbildung: Zinkfinger
![]() Jmol: Aktualisierter Zif268:DNA-Komplex Jmol14 (Java) | JSMol (HTML5)
Jmol: Aktualisierter Zif268:DNA-Komplex Jmol14 (Java) | JSMol (HTML5)
Zinkfingerproteine, von denen 900 im menschlichen Genom kodiert sind (einschließlich des oben beschriebenen eukaryotischen insulatorenbindenden Proteins CTCF), können mobilisiert werden, um bestimmte Mutationen in Zellen zu reparieren, was, wenn es in einem ausreichend hohen Prozentsatz der mutierten Zellen geschieht, bestimmte genetische Krankheiten wie einige Formen der schweren kombinierten Immunschwäche heilen könnte. Bei dieser neuen Technik (Urnov et al., 2005) werden mehrere verknüpfte Zn-Finger-Bindungsdomänen (eine der natürlich vorkommenden oder im Labor hergestellte mutierte Formen), die jeweils für eine bestimmte Nukleotidsequenz spezifisch sind, mit einer unspezifischen Endonuklease verbunden, die von dem Enzym FokI abgeleitet ist. Die Nuklease ist in dimerer Form aktiv, so dass der aktive Komplex zwei Endonuklease-Domänen benötigt, die jeweils an vier verschiedene Zn-Finger-Domänen gebunden sind, um sich an der Zielstelle zusammenzusetzen. Die Spezifität der Bindung wird durch die Selektion durch die Zn-Finger-Domänen erreicht. Die Nuklease macht dann eine Einkerbung in die DNA, und die Reparaturmechanismen der Wirtszelle setzen ein. Dieser Prozess umfasst die Strangtrennung, die homologe Rekombination der eingekerbten Region mit komplementärer DNA innerhalb der Zelle und die Reparatur der Einkerbung. Wird den Zellen überschüssige (nicht mutierte) Wildtyp-DNA zugesetzt und als Vorlage verwendet, würde die normale DNA-Reparatur-Mutation die Mutation reparieren. Urnov et al. haben gezeigt, dass bis zu 20 % der kultivierten Zellen, die eine Mutation enthalten, im Labor repariert werden können. Wenn diese Zellen einen selektiven Wachstumsvorteil erhalten, werden die mutierten Zellen schließlich durch Wildtypzellen ersetzt.
- Steroidhormonrezeptoren: (Eukaryoten) Im Gegensatz zu den meisten Hormonen, die an Zelloberflächenrezeptoren binden, passieren Steroidhormone (Cholesterinderivate) die Zellmembran und binden über eine Hormonbindungsdomäne an zytoplasmatische Rezeptoren. Dadurch verändert sich die Form des Rezeptors, der dann über eine DNA-Bindungsdomäne an eine bestimmte Stelle der DNA (Hormonreaktionselement) bindet. In einer Struktur, die dem Zinkfinger ähnelt, ist Zn 2+ tetraedrisch an 4 Cys koordiniert, in einer kugelförmigen Struktur, die als Dimer an zwei identische, aber umgekehrte DNA-Sequenzen (Palindrom) innerhalb der Hauptfurche bindet. (Beispiele von Palindromen: Fähig war ich, bevor ich Elba sah; Dennis und Edna speisen, sagte ich, als Enid und Edna sündigten.
Betrachten Sie den Glucocorticoid-Rezeptor (GR) als ein spezifisches Beispiel. Er bindet die DNA in Form eines Dimers. Die beiden DNA-Bindungsdomänen des Dimers assoziieren mit zwei benachbarten Hauptfurchen der DNA in der GR-Bindungssequenz (GBS), einer kurzen DNA-Sequenz innerhalb des Promotors. Meijsing et al. haben herausgefunden, dass die GBS nicht nur als Bindungsstelle für GR fungiert und die Transkription von Genen ermöglicht, sondern auch die Konformation des Rezeptors beeinflusst, wodurch die Gentranskription auf andere Weise reguliert wird. Die Gruppe konstruierte Luciferase-„Reportergene“, bei denen das GBS mit dem Gen für das Protein Luciferase verbunden ist und die das Protein Luciferase (das fluoresziert) exprimieren würden, wenn sie mit dem GBS transkribiert würden. Sie fanden heraus, dass die relative Transkriptionsaktivität nicht mit der relativen Bindungsaffinität von GR an den GBS korrelierte. GBS, die viel aktiver als andere waren, banden vergleichbar mit denen mit geringerer Aktivität, während GBS mit ähnlicher Transkriptionsaktivität mit unterschiedlicher Affinität banden. Dies zeigt, dass der GBS dem mit ihm verbundenen GR eine einzigartige Funktion verleiht (d. h. die Transkription wird nicht einfach dadurch beeinflusst, ob der GR an den GBS gebunden ist oder nicht). Es wurde festgestellt, dass ein �Hebelarm� des Rezeptors Konformationsänderungen erfährt, wenn er an DNA gebunden ist, wobei die Änderungen spezifisch für die Sequenz sind, an die er gebunden ist. Ein mutiertes Protein, GR-γ, wurde so hergestellt, dass es mit dem Wildtyp-Protein, GR-α, identisch ist, mit Ausnahme des Hebelarms, und es wurde festgestellt, dass es eine andere Transkriptionsaktivität hat, obwohl es an dieselbe Stelle der DNA bindet, was zeigt, dass der Hebelarm und seine Konformation die Transkription beeinflusst.
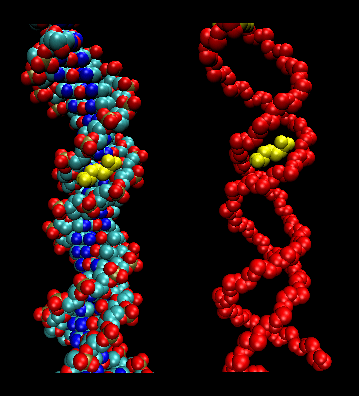
- Leucin-Reißverschlüsse (oder Scheren): (Eukaryoten) Diese Proteine enthalten Abschnitte von 35 Aminosäuren, in denen Leu wiederholt im Abstand von 7 Aminosäuren vorkommt. Diese Bereiche des Proteins bilden amphiphile Helices, mit Leu auf einer Seite und einem Leu nach zwei Windungen einer Helix. Zwei dieser Proteine können ein Dimer bilden, das durch die Bindung dieser unpolaren, leucinreichen amphiphilen Helices aneinander stabilisiert wird und eine gewundene Spirale bildet, ähnlich wie beim Muskelprotein Myosin. Der Leucin-Reißverschluss stellt die Proteinbindungsdomäne des Proteins dar. Die DNA-Bindungsdomäne befindet sich in den ersten 30 N-terminalen Aminosäuren, die basisch sind und eine Alpha-Helix bilden, wenn sich das Protein an die DNA bindet. Der Leucin-Reißverschluss dient dann dazu, zwei DNA-bindende Proteine zusammenzubringen, so dass die N-terminalen Basen-Helices basenspezifisch mit der Hauptfurche der DNA interagieren können. Valin und Isoleucin sind zusammen mit Leucin häufig in Aminosäureabschnitten zu finden, die interagieren können, um andere Arten von Spiralen zu bilden.
Abbildung: Leucin-Reißverschlüsse (erstellt mit VMD)
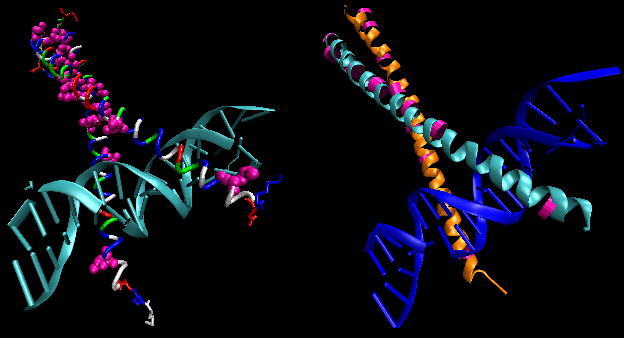
![]() Jmol: Aktualisierte Leucin-Zipper Jmol14 (Java) | JSMol (HTML5)
Jmol: Aktualisierte Leucin-Zipper Jmol14 (Java) | JSMol (HTML5)
Genauso wie Zinkfingernukleasen verwendet wurden, um die Reparatur von Mutationen zu induzieren, wurden in einer anderen Studie am Rattengenom speziell entwickelte ZFNs verwendet, um Brüche in ds-DNA zu verursachen, die Mutationen aus ungenauen DNA-Reparaturmechanismen (durch NHEJ) enthielten und daher spezifische Mutationen enthielten (Geurts, et al. 2009). Dieser Prozess, der „Knockout des Gens“, verhindert die Produktion des Proteins, das normalerweise von dem Zielgen transkribiert wird. Fünf- und Sechs-Finger-ZFNs wurden verwendet, um eine hohe Spezifität bei der gezielten Bindung an das Gen für drei verschiedene Proteine zu erreichen: grün fluoreszierendes Protein (GFP), Immunglobulin M (IgM) und Rab38. Der Knockout war bei 12 % der getesteten Ratten erfolgreich; diese Tiere hatten kein Wildtyp-Protein und keine Expression. Die ZFN waren ausreichend spezifisch, so dass keine Mutationen an einer der 20 vorhergesagten Nicht-Zielstellen beobachtet wurden. Diese Studie untermauert die Möglichkeit der Kontrolle von Transkription und Expression für die Behandlung von Krankheiten und die Bedeutung der spezifischen Bindung.
Wir haben gesehen, dass zwei Hauptfaktoren zur spezifischen Erkennung von DNA durch Proteine beitragen: die Bildung von Wasserstoffbrückenbindungen zu spezifischen Nukleotiddonatoren und -akzeptoren in der Hauptfurche und sequenzabhängige Verformungen der DNA-Helix zu veränderten Formen mit erhöhter Affinität von Proteinliganden. So kann beispielsweise das Tata-Bindungsprotein (TBP) mit einer verbreiterten Minor Grove in der TATA-Box interagieren. Neue Erkenntnisse belegen, dass Proteine darüber hinaus in der Lage sind, Informationen in Minor Grooves zu nutzen, die in Abhängigkeit von der Nukleotidsequenz „verengt“ wurden.
DNA-Spuren, die mit A angereichert sind, können zu verdrehten Konformationen führen, die Wasserstoffbrückenbindungen zwischen Basenpaaren in den Major Grooves verursachen, was zur Verengung der Minor Grooves führt. Hohe Mengen an AT-Basenpaaren sind in engen Nebenrillen (Breite <5,0 �) konzentriert, und CG-Basenpaare sind häufiger in breiten Nebenrillen zu finden.
Wie wirkt sich die Verengung der Nebenrillen auf die DNA-Erkennung aus? Schmale Nebenfurchen erhöhen das negative elektrostatische Potenzial der DNA, was sie zu einer spezifischeren und besser erkennbaren Stelle macht. Die Rückgratphosphate der DNA befinden sich näher an der Mitte der Rille, wenn diese schmal ist, so dass schmale Nebenrillen mit einem negativeren elektrostatischen Potenzial korrelieren.
Die mit der Nebenrille interagierenden Teile von Proteinen enthalten Arginin, dessen Seitenkette in der engeren und negativen Nebenrille untergebracht werden kann. Arginine können binden und sich in einigen Fällen als kurze Sequenzmotive einfügen, die die Spezifität der DNA-Formerkennung erhöhen. Arg wird gegenüber Lys bevorzugt, da die effektiven Radien der Ladung in Arg größer sind als die des Ladungsträgers in Lys. Dies würde zu einer geringeren Desolvationsenergie für Arg führen, was seine Bindung an die verengte Hauptfurche fördern würde. Diese Entdeckung zeigt, dass „die Rolle der DNA-Form bei der Annotation des gesamten Genoms und der Vorhersage von Transkriptionsfaktor-Bindungsstellen berücksichtigt werden muss“.
Abbildung: Arg in der T3c-Transposase-Bindung in der verengten Minor Grove des T3c-Transposons
- Gleitmodell für Protein/DNA-Interaktionen
Beitragende und Zuschreibungen
- Prof. Henry Jakubowski (College of St. Benedict/St. John’s University)