Einzel-Nukleotid-Polymorphismus
Einzel-Nukleotid-Polymorphismus (SNP) bezieht sich auf den Unterschied eines einzelnen Nukleotids an derselben Position in der genomischen DNA-Sequenz. Im Allgemeinen wird eine einzelne Nukleotidvariation mit einer Häufigkeit von mehr als 1 % als SNP bezeichnet. Bei SNPs handelt es sich nur um eine einzige Basenvariation, die durch einen einzigen Basenübergang oder eine Transversion verursacht werden kann. Im menschlichen Genom gibt es etwa einen SNP pro 1000 Basen, und die Gesamtzahl der SNPs im menschlichen Genom beträgt etwa 3 x 106.
SNP sind die dritte Generation genetischer Marker. Aufgrund ihrer Eigenschaften eignen sich SNPs besser als andere polymorphe Marker für Studien zur genetischen Pleiotropie bei komplexen Merkmalen und Krankheiten sowie zur populationsbasierten Generkennung. SNPs sind in der biomedizinischen Forschung von großer Bedeutung. SNPs wurden in genomweiten Assoziationsstudien als hochauflösende Marker bei der Genkartierung im Zusammenhang mit Krankheiten oder normalen Merkmalen verwendet. SNPs ohne erkennbare Auswirkung auf den Phänotyp (so genannte stille Mutationen) können aufgrund ihrer Menge und ihrer stabilen Vererbung über Generationen hinweg ebenfalls nützlich sein.
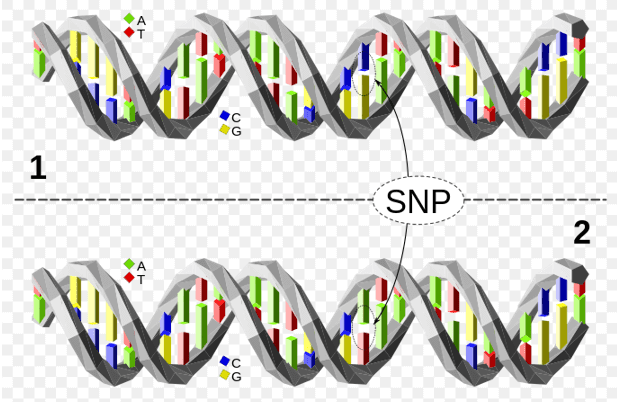 Abbildung 1. Das obere DNA-Molekül unterscheidet sich vom unteren DNA-Molekül an einer einzigen Basenpaar-Position (ein C/A-Polymorphismus).
Abbildung 1. Das obere DNA-Molekül unterscheidet sich vom unteren DNA-Molekül an einer einzigen Basenpaar-Position (ein C/A-Polymorphismus).
Microarray
Der DNA-Microarray bezeichnet einen Gen-Chip mit einer großen Anzahl von Sonden-DNA-Sequenzen in einer bestimmten Anordnung, die auf einem festen Substrat immobilisiert sind. Das Prinzip ist die Nukleinsäure-Hybridisierungstheorie, wobei die nachgewiesene Proben-DNA mit dem DNA-Mikroarray hybridisiert und verlängert wird. Anschließend werden die Fragmente der nicht-komplementären Bindungsreaktion auf dem Chip abgewaschen, und der Gen-Chip wird einem konfokalen Laserscan unterzogen. Die Intensitäten der Fluoreszenzsignale werden dann gemessen und durch eine bestimmte Datenverarbeitung als Häufigkeit der verschiedenen Gene interpretiert.
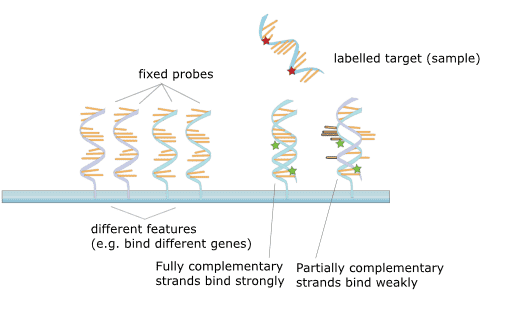 Abbildung 2. Das Prinzip des Microarrays.
Abbildung 2. Das Prinzip des Microarrays.
SNP-Microarray
SNP-Microarray verwendet bekannte Nukleotidsequenzen als Sonden, die mit den getesteten DNA-Sequenzen hybridisieren und eine qualitative und quantitative SNP-Analyse durch Signaldetektion ermöglichen. Im Vergleich zur traditionellen Einzelzelldiagnostik ist SNP-Microarray eine Methode mit hohem Durchsatz, mit der Tausende von Reaktionen auf der Oberfläche des Oligonukleotidchips gleichzeitig durchgeführt werden können.
Der Arbeitsablauf von SNP-Microarray
Der allgemeine Arbeitsablauf von SNP-Microarray ist in Abbildung 3 dargestellt. Kurz gesagt, gibt es mehrere Prozesse: SNP-Chip-Herstellung, Vorbereitung der genomischen DNA-Probe, Hybridisierung und Fluoreszenz-Scanning.
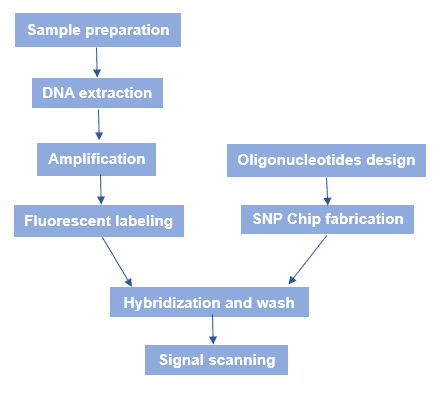 Abbildung 3. Der Arbeitsablauf eines SNP-Mikroarrays.
Abbildung 3. Der Arbeitsablauf eines SNP-Mikroarrays.
- Herstellung von SNP-Chips
Die vorgefertigten Oligonukleotid- oder cDNA-Chips werden geordnet und in hoher Dichte auf einem Glasträger angeordnet, um Mikroarrays oder Kernchips herzustellen. Zu den Herstellungsmethoden des Chips gehören die lichtgesteuerte In-situ-Synthese, die chemische Sprühmethode, die Kontaktpunktbeschichtungsmethode, die DNA-gesteuerte In-situ-Synthese, die berührungslose mikromechanische Druckmethode TOPSPOT® und die Softlithographie-Reproduktion usw. Insgesamt können nun 4×105 verschiedene DNA-Moleküle auf einem 1 cm2 großen Chip untergebracht werden. Die GeneChip®-Technologie von Affymetrix ermöglicht die Oligonukleotid-Synthese mit hoher Dichte in situ und ist ein Vorreiter auf diesem Gebiet. - Vorbereitung der genomischen DNA-Probe
Zunächst wird das Probenmaterial vorbereitet. Dann wird die genomische DNA extrahiert und durch PCR amplifiziert. Schließlich wird sie mit verschiedenen Fluoreszenzfarbstoffen markiert. - Hybridisierung und Scan
Die mit Fluoreszenz markierte genomische DNA wird unter geeigneten Reaktionsbedingungen mit dem SNP-Mikroarray hybridisiert. Nach dem Waschen wird ein spezieller Fluoreszenzscanner verwendet, um die Fluoreszenz zu scannen. Anschließend werden die Daten mit Hilfe der Computerbildanalyse verarbeitet, bevor die bioinformatische Analyse der Zielgene durchgeführt wird.
Das Ziel von CD Genomics ist es, zuverlässige SNP-Genotypisierungsdienste anzubieten, einschließlich Genotypisierung durch Sequenzierung, SNP-Mikroarray, MassARRAY SNP-Genotypisierung, Hi-SNPseq, SNaPshot Multiplex System für SNP-Genotypisierung und TaqMan SNP-Genotypisierung.
- Iwamoto K, Bundo M, Ueda J, et al. Detection of chromosomal structural alterations in single cells by SNP arrays: a systematic survey of amplification bias and optimized workflow. Plos One, 2007, 2(12): e1306.
- Ho C C, Mun K S, Naidu R. SNP array technology: an array of hope in breast cancer research. Malaysian Journal of Pathology, 2013, 35(1):33-43.
- Ahmad A, Iqbal M A. Significance of genome-wide analysis of copy number alterations and UPD in myelodysplastic syndromes using combined CGH-SNP arrays. Current Medicinal Chemistry, 2012, 19(22).
- Sund K L, Zimmerman S L, Thomas C, et al. Regions of homozygosity identified by SNP microarray analysis aid in the diagnosis of autosomal recessive disease and incidentally detect parental blood relationships. Genetics in Medicine Official Journal of the American College of Medical Genetics, 2013, 15(1):70-78.
- Liu S, Zhou Z, Lu J, et al. Generation of genome-scale gene-associated SNPs in catfish for the construction of a high-density SNP array. Bmc Genomics, 2011, 12(1):53.