Dada la relativa simplicidad estructural y repetitividad del ADN, se deduciría que las proteínas que se unen específicamente a él podrían tener motivos de dominio de unión al ADN comunes pero con cadenas laterales de aminoácidos específicas que permiten interacciones de unión específicas.
- Hélice-giro-hélice: se encuentra en las proteínas procariotas de unión al ADN.
Figura: hélice-giro-hélice
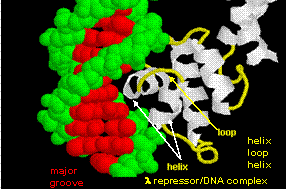
Las figuras muestran dos proteínas de este tipo, el represor cro del bacteriófago 434 y el represor lambda del bacteriófago lambda. (Los bacteriófagos son virus que infectan a las bacteias.) Nótese cómo la especificidad se consigue, en parte, por la formación de enlaces H específicos entre la proteína y el surco mayor del ADN operador.
Figura: Complejo Lambda Repressor/DNA
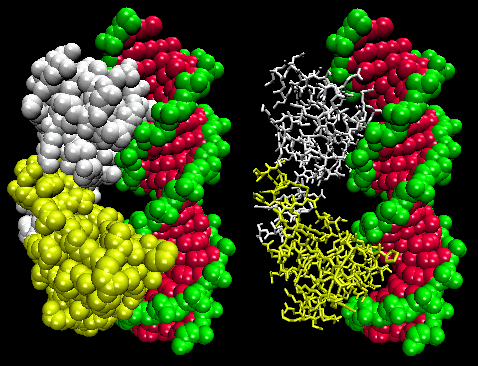
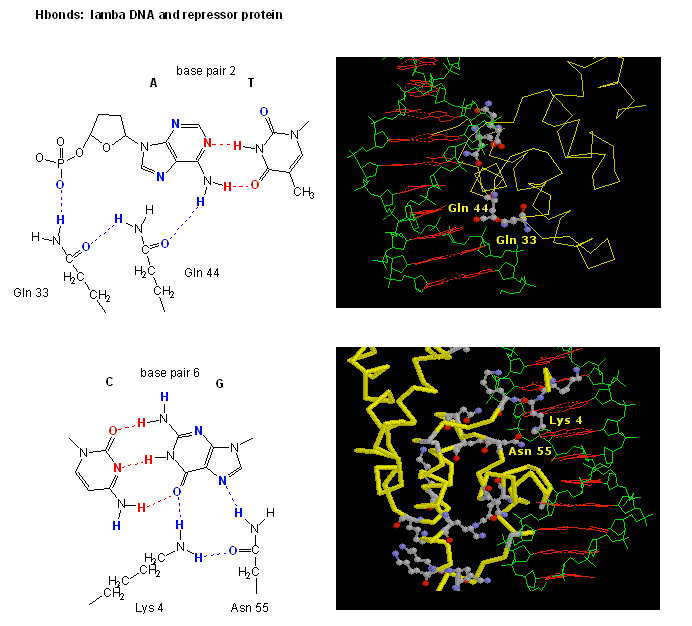
Figura: Interacciones de enlace H entre el represorλ y el ADN
![]() Jmol: Actualizado el complejo Lambda Repressor/DNA Jmol14 (Java) | JSMol (HTML5)
Jmol: Actualizado el complejo Lambda Repressor/DNA Jmol14 (Java) | JSMol (HTML5)
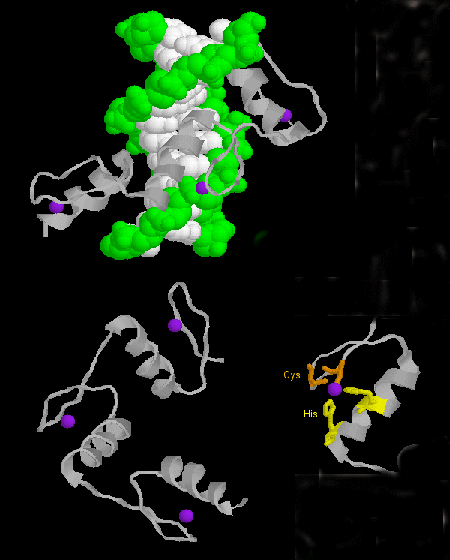
- dedo de zinc: (eucariotas) Estas proteínas tienen un motivo de secuencia común de X3-Cys-X2-4-Cys-X12-His-X3-4-His-X4- en el que X es cualquier aminoácido. El Zn2+ se coordina tetraédricamente con las cadenas laterales de Cys e His, que se encuentran en una de las dos cadenas beta antiparalelas y en una hélice alfa, respectivamente. El dedo de zinc, estabilizado por el zinc, se une al surco mayor del ADN. ]
Figura: dedo de zinc
![]() Jmol: Actualizado Zif268:DNA Complex Jmol14 (Java) | JSMol (HTML5)
Jmol: Actualizado Zif268:DNA Complex Jmol14 (Java) | JSMol (HTML5)
Las proteínas dedo de zinc, de las que se codifican 900 en el genoma humano (incluida la proteína eucariótica de unión a aislantes CTCF descrita anteriormente) pueden movilizarse para reparar realmente mutaciones específicas en las células, lo que, si se lleva a cabo en un porcentaje suficientemente alto de células mutantes, podría curar enfermedades genéticas específicas, como algunas formas de la enfermedad de inmunodeficiencia combinada grave. En esta nueva técnica (Urnov et al, 2005), múltiples dominios de unión de dedos de Zn enlazados, (uno de los naturales o formas mutantes producidas en el laboratorio), cada uno específico para una determinada secuencia de nucleótidos, se une a una endonucleasa no específica, derivada de la enzima FokI. La nucleasa es activa en forma dimérica, por lo que el complejo activo requiere dos dominios de endonucleasa, cada uno unido a cuatro dominios de dedos de Zn diferentes, para ensamblarse en el sitio objetivo. La especificidad de la unión se consigue mediante la selección de los dominios de dedo Zn. A continuación, la nucleasa hace una muesca en el ADN y se producen los mecanismos de reparación de la célula huésped. Este proceso implica la separación de la cadena, la recombinación homóloga de la región mellada con el ADN complementario dentro de la célula y la reparación de la mella. Si se añade un exceso de ADN de tipo salvaje (no mutado) a las células y se utiliza como plantilla, la mutación normal de reparación del ADN fijaría la mutación. Urnov et al han demostrado que hasta el 20% de las células cultivadas que contienen una mutación pueden ser reparadas en el laboratorio. Si estas células obtienen una ventaja selectiva de crecimiento, las células mutadas acabarían siendo sustituidas por células de tipo salvaje.
- receptores de hormonas esteroideas: (eucariotas) A diferencia de la mayoría de las hormonas, que se unen a los receptores de la superficie celular, las hormonas esteroides (derivados del colesterol) atraviesan la membrana celular y se unen a los receptores citoplasmáticos a través de un dominio de unión a la hormona. Esto cambia la forma del receptor, que luego se une a un sitio específico en el ADN (elemento de respuesta hormonal) a través de un dominio de unión al ADN. En una estructura análoga a la del dedo de zinc, el Zn 2+ se coordina tetraédricamente con 4 Cys, en una estructura de tipo globular que se une como dímero a dos secuencias idénticas pero invertidas de ADN (palíndromo) dentro del surco mayor. (Ejemplos de palíndromos: Capaz era yo antes de ver a Elba; Dennis y Edna cenan, dije yo, mientras Enid y Edna pecan.
Considere el receptor de glucocorticoides (GR) como un ejemplo específico. Se une el ADN como un dímero. Los dos dominios de unión al ADN del dímero se asocian con dos ranuras principales adyacentes del ADN en la secuencia de unión al GR (GBS), una secuencia corta de ADN dentro del promotor. Meijsing y otros han descubierto que la GBS no sólo actúa como sitio de unión del GR, permitiendo la transcripción de los genes, sino que también afecta a la conformación del receptor, haciendo que la transcripción de los genes se regule de otra manera. El grupo construyó «genes reporteros» de luciferasa que tienen el GBS unido al gen de la proteína luciferasa, que expresaría la proteína luciferasa (que es fluorescente) si se transcribiera, con el GBS. Descubrieron que la actividad transcripcional relativa no se correlacionaba con la afinidad de unión relativa del GR al GBS. Los GBS que eran mucho más activos que otros se unían de forma comparable a los de menor actividad, mientras que los GBS con una actividad transcripcional similar se unían con afinidades diferentes. Esto demuestra que el GBS confiere una función única al GR asociado a él (es decir, la transcripción no se ve simplemente afectada por el hecho de que el GR esté o no unido al GBS). Se descubrió que un �brazo de palanca� del receptor experimentaba cambios de conformación cuando se unía al ADN, con cambios específicos para la secuencia a la que se unía. Se hizo que una proteína mutante, GR-γ, fuera idéntica a la proteína de tipo salvaje, GR-α, excepto en el brazo de palanca se encontró que tenía una actividad transcripcional diferente a pesar de que se estaban uniendo al mismo sitio en el ADN, lo que demuestra que el brazo de palanca y su conformación afecta a la transcripción.
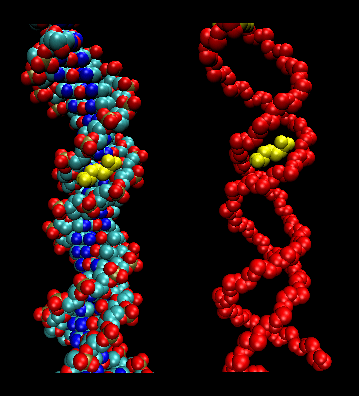
- cremalleras (o tijeras) de leucina: (eucariotas) Estas proteínas contienen tramos de 35 aminoácidos en los que la Leu se encuentra repetidamente a intervalos de 7 aminoácidos. Estas regiones de la proteína forman hélices anfifílicas, con Leu en una cara, una Leu después de dos vueltas de hélice. Dos de estas proteínas pueden formar un dímero, estabilizado por la unión de estas hélices anfifílicas no polares y ricas en leucina entre sí, formando un espiral, como en la proteína muscular miosina. La cremallera de leucina representa el dominio de unión de la proteína. El dominio de unión al ADN se encuentra en los primeros 30 aminoácidos N-terminales, que son básicos y forman una hélice alfa cuando la proteína se une al ADN. La cremallera de leucina funciona entonces para unir dos proteínas de unión al ADN, permitiendo que las hélices de las bases N-terminales interactúen con el surco mayor del ADN de forma específica para cada base. La valina y la isoleucina, junto con la leucina, se encuentran a menudo en tramos de aminoácidos que pueden interactuar para formar otros tipos de espirales.
Figura: cremalleras de leucina (realizadas con VMD)
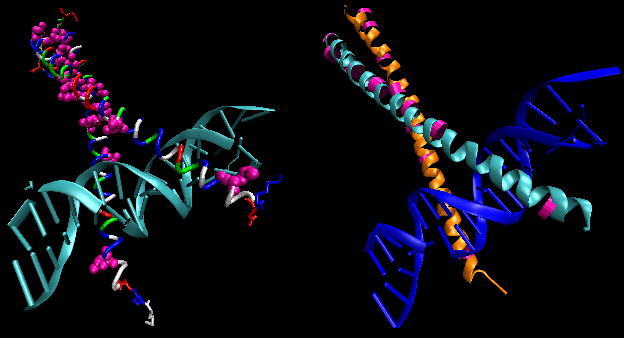
![]() Jmol: Cremalleras de leucina actualizadas Jmol14 (Java) | JSMol (HTML5)
Jmol: Cremalleras de leucina actualizadas Jmol14 (Java) | JSMol (HTML5)
Al igual que las nucleasas de dedos de zinc se han utilizado para inducir la reparación de mutaciones, en otro estudio del genoma de la rata se utilizaron ZFNs especialmente diseñados para provocar roturas en el ds-ADN que contenían mutaciones de un mecanismo de reparación del ADN inexacto (por NHEJ) y que, por tanto, contenían mutaciones específicas (Geurts, et al. 2009). Este proceso, �knockout del gen,� impide la producción de la proteína normalmente transcrita por el gen objetivo. Se utilizaron ZFN de cinco y seis dedos para lograr un alto nivel de especificidad en la unión dirigida al gen para tres proteínas diferentes: la proteína verde fluorescente (GFP), la inmunoglobulina M (IgM) y Rab38. El knockout tuvo éxito en el 12% de las ratas analizadas; estos animales no tenían la proteína de tipo salvaje y no tenían expresión. Los ZFN fueron lo suficientemente específicos como para que no se observaran mutaciones en ninguno de los 20 sitios no objetivo previstos. Este estudio apoya la viabilidad del control de la transcripción y la expresión para el tratamiento de enfermedades y la importancia de la unión específica.
Hemos visto que hay dos factores principales que contribuyen al reconocimiento específico del ADN por parte de las proteínas; la formación de enlaces de hidrógeno a donantes y aceptores de nucleótidos específicos en el surco principal, y las deformaciones dependientes de la secuencia de la hélice del ADN a formas alteradas con mayor afinidad de los ligandos de las proteínas. Por ejemplo, la proteína de unión a Tata (TBP) puede interactuar con un surco menor ensanchado en la caja TATA. Nuevos descubrimientos apoyan que además las proteínas son capaces de utilizar la información de los surcos menores que se han «estrechado» dependiendo de la secuencia de nucleótidos.
Los surcos de ADN enriquecidos en A pueden dar lugar a conformaciones de torsión que provocan la unión de hidrógeno entre pares de bases en los surcos mayores, lo que resulta en el estrechamiento de los surcos menores. En los surcos menores estrechos se concentran grandes cantidades de pares de bases AT (anchura <5,0 �) y los pares de bases CG se encuentran con más frecuencia en los surcos menores anchos.
¿Cómo afecta el estrechamiento del surco menor al reconocimiento del ADN? Los surcos menores estrechos aumentan el potencial electrostático negativo del ADN, convirtiéndolo en un sitio más específico y reconocible. Los fosfatos de la columna vertebral del ADN están más cerca del centro del surco cuando éste es estrecho, correlacionando así los surcos menores estrechos con un potencial electrostático más negativo.
Las partes de las proteínas que interactúan en el surco menor contienen arginina cuya cadena lateral puede acomodarse en el surco menor más estrecho y negativo. Las argininas pueden unirse y, en algunos casos, insertarse como motivos de secuencias cortas que mejoran la especificidad del reconocimiento de la forma del ADN. Se prefiere Arg sobre Lys ya que los radios efectivos de la carga en Arg son mayores que los del portador de carga en Lys. Esto llevaría a una disminución de la energía de desolvatación para Arg, lo que promovería su unión al surco mayor estrechado. Este descubrimiento demuestra que «el papel de la forma del ADN debe tenerse en cuenta a la hora de anotar todo el genoma y predecir los sitios de unión a factores de transcripción».
Figura: Arg en la unión de la transposasa T3c en la arboleda menor estrecha del transposón T3c
- Modelo de deslizamiento para las interacciones proteína/ADN
Contribuidores y atribuciones
- Profesor Henry Jakubowski (College of St. Benedict/St. John’s University)
.