Editorial sobre el Tema de Investigación
El Papel de las Proteínas AAA+ en Reparación y Degradación de Proteínas
Las ATPasas Asociadas a Diversas Actividades Celulares (AAA+) comprenden una superfamilia de proteínas que realizan una gran variedad de funciones esenciales para la fisiología celular, incluyendo el control de la homeostasis de las proteínas, la replicación del ADN, la recombinación, la remodelación de la cromatina, el procesamiento del ARN ribosómico, la orientación molecular, la biogénesis de los orgánulos y la fusión de las membranas (Hanson y Whiteheart, 2005; Erzberger y Berger, 2006; Snider et al., 2008). Los miembros de esta superfamilia se definen por la presencia de lo que se denomina el dominio AAA+ que contiene los motivos canónicos Walker A y B necesarios para la unión e hidrólisis del ATP (Hanson y Whiteheart, 2005). Normalmente, los genomas codifican entre diez y varios centenares de miembros de la familia AAA+ (Tabla 1; Finn et al., 2017), cada uno de los cuales se cree adaptado a nichos funcionales específicos que requieren mecanismos precisos de reconocimiento y procesamiento de sustratos (Hanson y Whiteheart, 2005). La sorprendente radiación adaptativa de las proteínas AAA+ para operar en diversos entornos ilustra la versátil utilidad del dominio AAA+ (Erzberger y Berger, 2006). Las proteínas AAA+ suelen formar complejos hexaméricos y actúan como motores para remodelar otras proteínas, ADN/ARN o complejos multicomponentes (Figura 1). De hecho, muchas chaperonas y proteasas dependientes de ATP son o tienen subunidades que pertenecen a esta superfamilia (Figura 1; Olivares et al., 2016).
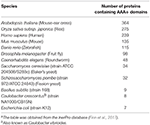
Tabla 1. Número de proteínas AAA+ en organismos modeloa.
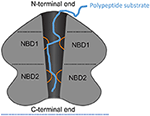
Figura 1. Esquema de ClpA como ejemplo de hexámero AAA+. Representación esquemática de la arquitectura de dominios e interacciones de ClpA como ejemplo de hexámero AAA+ con dos dominios AAA+ por monómero. Aquí se muestra una vista lateral recortada del hexámero ClpA para representar el canal central conductor de polipéptidos. ClpA contiene tres dominios, incluyendo un dominio N-terminal y dos dominios AAA+: dominio de unión a nucleótidos 1 y 2 (NBD1 y NBD2). El dominio N-terminal interactúa con reguladores de la especificidad del sustrato, mientras que el extremo C-terminal interactúa con la proteasa de cámara ClpP. Los bucles móviles de NBD1 y NBD2 (naranja) se proyectan en el canal central y enganchan al sustrato polipeptídico (azul) permitiendo así el acoplamiento de la hidrólisis de ATP a la translocación del polipéptido a través del canal central.
En los últimos años, se ha producido un progreso sustancial en la identificación de la estructura y el mecanismo funcional de un gran número de proteínas AAA+ (Gates et al., 2017; Puchades et al., 2017; Ripstein et al., 2017; Zehr et al., 2017). En este tema de investigación, se transmiten varios elementos de este emocionante progreso en 21 artículos, que abarcan una visión estructural y mecanística detallada de varias chaperonas y proteasas AAA+, entre ellas: ClpX (Alhuwaider y Dougan; Bittner et al.; Elsholz et al.; LaBreck et al.; Vass et al.), ClpA (Bittner et al.; Duran et al.), ClpB y Hsp104 (Chang et al.Duran et al.; Franke et al.; Johnston et al.), Hsp78 (Abrahão et al.), ClpC (Alhuwaider y Dougan; Elsholz et al.), ClpE (Elsholz et al.), Pontin (Mao y Houry), Reptin (Mao y Houry), FtsH (Alhuwaider y Dougan), 19S proteasoma (Snoberger et al.; Yedidi et al.), Lon (Alhuwaider y Dougan; Bittner et al.; Fishovitz et al.), p97 (Hänzelmann y Schindelin; Saffert et al.; Ye et al.), Pex1/6 (Saffert et al.), CbbQ (Mueller-Cajar), rubisco activasa (Bhat et al.), torsinas (Chase et al.) y proteasas mitocondriales AAA+ (Glynn). A continuación, presentamos estos fascinantes trabajos.
Estudios sobre ClpXP, Lon y proteasas dependientes de ATP relacionadas
En su artículo de investigación, «The Protein Chaperone ClpX Targets Native and Non-native Aggregated Substrates for Remodeling, Disassembly, and Degradation with ClpP,» LaBreck et al. realizan una serie de elegantes experimentos para establecer que ClpX posee actividad desagregadora contra polipéptidos que contienen señales específicas de reconocimiento de ClpX (LaBreck et al.). En presencia de ClpP, ClpX acopla la desagregación de estos sustratos a su degradación. Es importante destacar que también establecen que ClpXP impide la acumulación de agregados formados por proteínas que llevan señales de reconocimiento de ClpX in vivo (LaBreck et al.). Estos estudios ponen de manifiesto que ClpX es una desagregadora de proteínas, algo que hasta ahora se había subestimado.
En su artículo de investigación, «The Essential Role of ClpXP in Caulobacter crescentus Requires Species Constrained Substrate Specificity» (El papel esencial de ClpXP en Caulobacter crescentus requiere una especificidad de sustrato restringida por la especie), Vass et al. exploran las funciones específicas de ClpX por especie (Vass et al.). Curiosamente, ClpX es esencial en algunas especies como C. crescentus, pero no lo es en otras bacterias como E. coli (Vass et al.). Es importante destacar que E. coli ClpX fue incapaz de complementar a C. crescentus ClpX in vivo (Vass et al.). Esta falta de actividad se debió a las diferencias específicas de la especie en el dominio N-terminal de ClpX, que son críticas para el procesamiento de la subunidad cargadora de la abrazadera de replicación DnaX en C. crescentus. Así, las pequeñas diferencias en la especificidad de ClpX pueden ser particularmente críticas para especies bacterianas específicas.
En su revisión sobre «Diversidad funcional de los complejos de proteasas AAA+ en Bacillus subtilis», Elsholz et al. discuten las funciones de varias proteasas AAA+ en B. subtilis, a saber: ClpCP, ClpEP, ClpXP, ClpYQ, LonA/B y FtsH (Elsholz et al.). En ellos se discute cómo las diferentes respuestas al estrés controlan su expresión y los fenotipos observados tras la supresión de estas diferentes proteasas. Se describe la capacidad de algunas de estas proteasas para controlar la competencia, la esporulación, la motilidad y la formación de biopelículas. Por último, los autores discuten la posibilidad de utilizar estas proteasas para el desarrollo de nuevos antibióticos.
En su revisión titulada «AAA+ Machines of Protein Destruction in Mycobacteria», Alhuwaider et al. (Alhuwaider y Dougan) discuten los recientes avances en la determinación de la estructura y función de las proteasas AAA+ de las micobacterias. Estas proteasas son: ClpXP1P2, ClpC1P1P2, Lon, FtsH y Mpa. Los autores también hablan del sistema Pup-proteasoma (PPS) presente en las micobacterias, que es equivalente al sistema ubiquitina-proteasoma de los eucariotas. Alhuwaider et al. concluyen con un análisis de nuevos compuestos que desregulan o inhiben la actividad de ClpP1P2 y otros que desregulan ClpC1. Estos compuestos tienen actividades prometedoras contra las micobacterias.
En un artículo de investigación titulado «The Copper Efflux Regulator CueR Is Subject to ATP-Dependent Proteolysis in Escherichia coli», Bittner et al. demuestran que las proteasas AAA+ Lon, ClpXP y ClpAP son responsables de la degradación de E. coli CueR, que es un factor de transcripción que controla la inducción del sistema Cue de eflujo de cobre (Bittner et al.). Los autores descubrieron que el reconocimiento de CueR por parte de las proteasas AAA+ requiere el terminal C accesible de CueR. Concluyen que las proteasas dependientes de ATP son necesarias para la homeostasis del cobre en E. coli.
Fishovitz et al. realizan una comparación detallada entre la Lon humana y la de E. coli en su artículo de investigación titulado «Utilization of Mechanistic Enzymology to Evaluate the Significance of ADP Binding to Human Lon Protease» (Fishovitz et al.). Mediante un estudio mecanístico detallado, descubrieron que, a diferencia de la Lon de E. coli, la Lon humana tiene una baja afinidad por el ADP a pesar de mostrar valores de kcat y KM comparables en la actividad ATPasa. Proponen que la Lon humana no está regulada por un mecanismo de intercambio ADP/ATP promovido por el sustrato. Estas diferencias entre la Lon humana y la de E. coli podrían permitir el desarrollo futuro de inhibidores de Lon específicos para cada especie.
En su revisión sobre «Proteasas AAA mitocondriales multifuncionales», el Dr. Glynn analiza las dos proteasas AAA mitocondriales, i-AAA y m-AAA (Glynn). Ambas son proteínas de la membrana interna mitocondrial. Sin embargo, la i-AAA proyecta los dominios ATPasa y proteasa en el espacio intermembranal mitocondrial, mientras que la proteasa m-AAA proyecta los dominios catalíticos en la matriz. Se discuten las estructuras de estas proteasas, así como su mecanismo de funcionamiento. Las proteasas pueden llevar a cabo la degradación completa de sustratos, pero también pueden escindir sólo ciertos sustratos, como en el caso de MrpL32 y Atg32.
ClpB y Hsp104
En su mini-revisión, «Structural Elements Regulating AAA+ Protein Quality Control Machines», Chang et al. discuten cómo el bucle de poro-1, el motivo de señalización entre subunidades y el motivo de inserción del presensor I podrían contribuir a la actividad de dos desagregadoras de Hsp100, la ClpB bacteriana y la Hsp104 de levadura (Chang et al.). Proponen un modelo sobre cómo estos elementos estructurales podrían permitir que el ciclo de la ATPasa AAA+ se acople a la translocación de sustratos a través del canal central de ClpB y Hsp104. Se cree que este proceso de translocación de polipéptidos apuntala la forma en que ClpB y Hsp104 extraen los polipéptidos de las estructuras agregadas (Chang et al.).
Duran et al. proporcionan un «Análisis comparativo de la estructura y función de los motores AAA+ ClpA, ClpB y Hsp104: hilos comunes y funciones dispares» en su revisión (Duran et al.). En él discuten la capacidad de estas tres proteínas AAA+ (ClpA, ClpB y Hsp104) para translocar polipéptidos a través de sus complejos hexaméricos. Todas estas proteínas tienen dos dominios AAA+ y se sabe que despliegan proteínas. Además, se sabe que ClpB y Hsp104 funcionan como desagregadoras, mientras que ClpA puede formar un complejo con la proteasa ClpP. Los autores destacan la necesidad de utilizar métodos cinéticos de estado transitorio para examinar los mecanismos cinéticos de estas proteínas motoras. Describen cómo el uso de tales métodos les permitió demostrar que, por ejemplo, ClpA transloca polipéptidos a unos 20 aa s-1, mientras que en complejo con la proteasa ClpP, la velocidad de translocación de ClpA es aún mayor, de unos 35 aa s-1. Los autores también discuten la importancia de la chaperona Hsp70 en la función de ClpB/Hsp104,y la observación de la especificidad de especie en la interacción entre Hsp70 y ClpB/Hsp104.
En su artículo de investigación titulado «Mutant Analysis Reveals Allosteric Regulation of ClpB Disaggregase» (El análisis de mutantes revela la regulación alostérica de la desagregadora ClpB), Franke et al. llevan a cabo un análisis mutacional de la desagregadora ClpB de E. coli para caracterizar su regulación alostérica (Franke et al.). ClpB puede dividirse en un dominio N-terminal y dos dominios AAA+ separados por una región helicoidal denominada dominio M. Los autores identifican un residuo altamente conservado en el primer dominio AAA+, A328. Se descubrió que el mutante ClpB-A328V tenía una actividad ATPasa muy elevada y presentaba toxicidad celular. Inesperadamente, la elevada actividad ATPasa de ClpB-A328V se debía principalmente al segundo anillo AAA+, según se evaluó mediante espectrometría de masas de intercambio de hidrógeno de amidas. Los autores concluyen que A328 es un residuo crucial en el control de la hidrólisis de ATP en ambos anillos AAA+ de ClpB.
En su artículo de investigación titulado «Substrate Discrimination by ClpB and Hsp104», Johnston et al. describen las preferencias innatas de sustrato de ClpB y Hsp104 en ausencia de los sistemas de chaperonas DnaK y Hsp70 (Johnston et al.). Demuestran que la especificidad del sustrato viene determinada por el primer dominio AAA+ de cada proteína. Llegaron a esta conclusión probando la capacidad de las dos chaperonas para actuar sobre varios sustratos modelo. También probaron diferentes quimeras de las dos chaperonas.
En «Hsp78 (78 kDa Heat Shock Protein), un miembro representativo de la familia AAA que se encuentra en la matriz mitocondrial de Saccharomyces cerevisiae», Abrahão et al. discuten la estructura y función de Hsp78 (Abrahao et al.). Hsp78 es el parálogo mitocondrial de Hsp104, que funciona en la desagregación y reactivación de proteínas (Abrahao et al.). Curiosamente, Hsp104 y Hsp78 se perdieron en la transición de protozoos a metazoos (Abrahao et al.). Sin embargo, Abrahao et al. discuten la existencia de ANKCLP, que aparece junto a Hsp78 y Hsp104 en los protozoos y sobrevive a la transición evolutiva a los metazoos. ANKCLP posee un dominio AAA+ similar al dominio de unión a nucleótidos 2 (NBD2) de Hsp104 y Hsp78, pero por lo demás es muy divergente. Curiosamente, las mutaciones en ANKCLP causan aciduria 3-metilglutacónica, atrofia cerebral progresiva, discapacidad intelectual, neutropenia congénita, cataratas y trastornos del movimiento en humanos (Abrahao et al.).
p97
En su revisión, «Structure and Function of p97 and Pex1/6 Type II AAA+ Complexes» (Estructura y función de los complejos AAA+ de p97 y Pex1/6 de tipo II), Saffert et al. discuten dos complejos AAA+ diferentes que remodelan las proteínas sustrato ubiquitinadas (Saffert et al.). Una de las funciones de p97 es dislocar sustratos ubiquitinados de la membrana del RE al proteasoma durante la degradación asociada al RE (Saffert et al.). Por el contrario, Pex1/Pex6 es un motor heterohexamérico compuesto por subunidades alternas de Pex1 y Pex6, que es esencial para la biogénesis y la función de los peroxisomas. Se discuten las estructuras recientes de p97 y Pex1/6 mediante criomicroscopía electrónica y se destacan las diferencias estructurales clave.
En su revisión titulada «A Mighty ‘Protein Extractor’ of the Cell: Structure and Function of the p97/CDC48 ATPase», Ye et al. resumen los conocimientos actuales sobre la estructura y la función de p97 y su papel en varias enfermedades (Ye et al.). p97 tiene dos dominios AAA+ conectados con un enlace corto. También tiene un dominio N-terminal, que media sus interacciones con diferentes proteínas adaptadoras. Los autores ofrecen un análisis detallado de la estructura de p97 y del efecto de los nucleótidos en sus diferentes conformaciones. Estos estudios se basan en el uso de técnicas como la EM, la cristalografía de rayos X y la microscopía de fuerza atómica de alta velocidad. A continuación, los autores discuten las funciones multicelulares de esta proteína altamente conservada, incluyendo su papel en la degradación de proteínas asociada al RE (ERAD), la degradación asociada a la mitocondria (MAD) mediante la extracción de polipéptidos de la membrana externa mitocondrial, y la degradación asociada al ribosoma (RAD). Por último, Ye et al. ofrecen un resumen de las mutaciones de p97 que conducen a varias enfermedades humanas como la IBMPFD (miopatía de cuerpos de inclusión asociada a la enfermedad ósea de Paget y a la demencia frontotemporal)], la FALS (esclerosis lateral amiotrófica familiar), la CMT2Y (enfermedad de Charcot-Marie-Tooth, tipo 2Y), las paraplejias espásticas hereditarias (HSP), la enfermedad de Parkinson (EP) y la enfermedad de Alzheimer (EA).
En su revisión sobre p97 titulada «The Interplay of Cofactor Interactions and Post-translational Modifications in the Regulation of the AAA+ ATPase p97», Hänzelmann y Schindelin discuten cómo diferentes cofactores modulan la actividad de la ATPasa p97 (Hanzelmann y Schindelin). Destacan el hecho de que la capacidad de p97 de participar en un gran número de procesos celulares se debe al gran número de cofactores que interactúan con esta proteína. Los autores han identificado tres clases diferentes de cofactores de p97, a saber (i) Cofactores reclutadores de sustratos, como las proteínas UBA-UBX y UFD1-NPL4, (ii) Cofactores procesadores de sustratos, como las ubiquitinas (E3) ligasas y las deubiquitinasas (DUBs), y (iii) Cofactores reguladores, como las proteínas UBX, que pueden secuestrar o reciclar hexámeros de p97. Los autores también analizan el papel de las modificaciones postraduccionales en la actividad de p97 y en sus interacciones con sus cofactores y sustratos.
Proteínas AAA+ del proteasoma
En «AAA-ATPasas en la degradación de proteínas», Yedidi et al. revisan las actividades de Rpt1, Rpt2, Rpt3, Rpt4, Rpt5 y Rpt6, que son las ATPasas AAA+ del proteasoma eucariótico, así como de algunos de sus parientes bacterianos como PAN, Mpa y VAT (Yedidi et al.). Se centran en las nuevas tecnologías para entender cómo funcionan estas AAA+ ATPasas translocando polipéptidos desplegados a la cámara proteolítica de la proteasa (Yedidi et al.). Los cambios conformacionales dentro del anillo AAA+ y la cámara adyacente de la proteasa parecen generar un mecanismo de bombeo peristáltico para entregar sustratos para su degradación (Yedidi et al.).
En su artículo de investigación, «Las ATPasas proteasomales utilizan una estrategia lenta pero altamente procesal para desplegar proteínas», Snoberger et al. establecen que las proteínas AAA+ proteasomales emplean un mecanismo motor de baja velocidad pero altamente procesivo para entregar sustratos a la cavidad proteolítica del proteasoma (Snoberger et al.). Este mecanismo contrasta con ClpX, que utiliza un mecanismo motor de alta velocidad pero menos procesivo para entregar sustratos a la proteasa ClpP para su degradación. Estas diferencias en el mecanismo motor pueden haber evolucionado en respuesta a las diferentes demandas de su clientela específica.
Rubisco Activasas
En su revisión sobre «Rubisco Activasas: AAA+ Chaperones Adapted to Enzyme Repair» Bhat et al. discuten la función única de la Rubisco activasa (Rca) en la remodelación de la Rubisco (Bhat et al.). La Rca es una chaperona AAA+ muy conservada en los organismos fotosintéticos, desde las bacterias hasta las plantas superiores. Rubisco es la enzima Ribulosa-1,5-bisfosfato carboxilasa/oxigenasa, que participa en la fijación del CO2 atmosférico durante la fotosíntesis. Es la proteína más abundante en la Tierra y es la enzima clave en la síntesis de toda la materia orgánica del planeta. Sin embargo, la Rubisco es una enzima pobre y es fácilmente inhibida por los productos secundarios de sus reacciones catalíticas o por compuestos sintetizados por algunas plantas en condiciones de poca luz. La función de la Rca es aliviar o «curar» a la Rubisco de tales inhibiciones problemáticas. Los autores discuten la estructura de Rca de diferentes especies y los posibles mecanismos de su función.
El Dr. Mueller-Cajar ofrece una revisión sobre «Las diversas máquinas AAA+ que reparan los sitios activos de Rubisco inhibidos» (Mueller-Cajar). Discute la presencia de tres clases evolutivamente distintas de Rubisco activasas (Rcas): (1) Rcas de tipo verde y (2) de tipo rojo que se encuentran principalmente en eucariotas fotosintéticos del linaje de los plástidos verdes y rojos, respectivamente, y (3) CbbQO presente en bacterias quimioautotróficas. Discute la evolución de estas activasas y su posible uso en biología sintética para mejorar la actividad de la Rubisco en las plantas.
Torsin
En su artículo de perspectiva, «Torsin ATPases: Harnessing Dynamic Instability for Function», Chase et al. hablan de las Torsinas, que también están relacionadas filogenéticamente con la NBD2 de la Hsp104 de la levadura (Chase et al.). Las torsinas son las únicas ATPasas AAA+ localizadas dentro del RE y de la envoltura nuclear conectada (Chase et al.). Curiosamente, las mutaciones en TorsinA causan la distonía DYT1, un trastorno neurológico en humanos (Chase et al.). Las torsinas exhiben una débil actividad ATPasa que se incrementa a través de la complementación del sitio activo debido al coensamblaje con cofactores accesorios específicos LAP1 y LULL1 (Chase et al.). Chase et al. sugieren que el ensamblaje y el desensamblaje dinámicos de los complejos Torsin/cofactores desempeñan papeles importantes en su función en el tráfico nuclear y en el ensamblaje del complejo nuclear-poroso (Chase et al.).
Pontin y Reptin
En su extensa revisión sobre «El papel de Pontin y Reptin en la fisiología celular y la etiología del cáncer», Mao y Houry discuten las múltiples funciones de las ATPasas AAA+ altamente conservadas de Pontin y Reptin (Mao y Houry). Estas dos proteínas suelen funcionar juntas como un complejo, pero también pueden funcionar de forma independiente. Los autores destacan las funciones de Pontin y Reptin en la remodelación de la cromatina. También discuten cómo Pontin y Reptin modulan las actividades transcripcionales de varios protooncogenes como MYC y β-catenina. Mao y Houry dilucidan cómo se ha descubierto que Pontin y Reptin son necesarias para el ensamblaje de los complejos de señalización PIKK, así como de la telomerasa, el huso mitótico, la ARN polimerasa II y las snoRNPs. Los autores concluyen con una visión general de los esfuerzos actuales dirigidos a identificar inhibidores de Pontin y Reptin para ser desarrollados como nuevos anticancerígenos.
Observaciones finales
En conclusión, esta colección de 21 artículos destaca una serie de importantes aspectos estructurales y mecanísticos de las proteínas AAA+ implicadas en la reparación y degradación de proteínas. Estamos entusiasmados por ver cómo el campo continuará desarrollándose durante la actual revolución de la crio-EM (Egelman, 2016). Anticipamos que la crio-EM permitirá una comprensión más profunda de cómo funcionan estas fascinantes máquinas moleculares en diversas situaciones (Gates et al., 2017; Puchades et al., 2017; Ripstein et al., 2017; Zehr et al., 2017).
Contribuciones de los autores
Todos los autores enumerados han realizado una contribución sustancial, directa e intelectual al trabajo, y lo han aprobado para su publicación.
Declaración de conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un potencial conflicto de intereses.
Agradecimientos
El trabajo en el laboratorio de WH en esta área está financiado por una subvención del Proyecto de Investigación de los Institutos Canadienses de Salud (PJT-148564). JS cuenta con el apoyo de la subvención R01GM099836 de los NIH.
Egelman, E. H. (2016). La revolución actual en Cryo-EM. Biophys. J. 110, 1008-1012. doi: 10.1016/j.bpj.2016.02.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Erzberger, J. P., y Berger, J. M. (2006). Relaciones evolutivas y mecanismos estructurales de las proteínas AAA+. Annu. Rev. Biophys. Biomol. Struct. 35, 93-114. doi: 10.1146/annurev.biophys.35.040405.101933
PubMed Abstract | CrossRef Full Text | Google Scholar
Finn, R. D., Attwood, T. K., Babbitt, P. C., Bateman, A., Bork, P., Bridge, A. J., et al. (2017). InterPro en 2017: más allá de las anotaciones de familias y dominios de proteínas. Nucleic Acids Res. 45, D190-D199. doi: 10.1093/nar/gkw1107
PubMed Abstract | CrossRef Full Text | Google Scholar
Gates, S. N., Yokom, A. L., Lin, J., Jackrel, M. E., Rizo, A. N., Kendsersky, N. M., et al. (2017). Mecanismo de translocación de polipéptidos tipo trinquete de la desagregación AAA+ Hsp104. Science 357, 273-279. doi: 10.1126/science.aan1052
PubMed Abstract | CrossRef Full Text | Google Scholar
Hanson, P. I., y Whiteheart, S. W. (2005). Proteínas AAA+: si tienen motor, funcionarán. Nat. Rev. Mol.Cell Biol. 6, 519-529. doi: 10.1038/nrm1684
PubMed Abstract | CrossRef Full Text | Google Scholar
Olivares, A. O., Baker, T. A., y Sauer, R. T. (2016). Perspectivas mecanísticas de las proteasas AAA+ bacterianas y de las máquinas de remodelación de proteínas. Nat. Rev. Microbiol. 14, 33-44. doi: 10.1038/nrmicro.2015.4
PubMed Abstract | CrossRef Full Text | Google Scholar
Puchades, C., Rampello, A. J., Shin, M., Giuliano, C. J., Wiseman, R. L., Glynn, S. E., et al. (2017). La estructura de la proteasa AAA+ de la membrana interna mitocondrial YME1 permite comprender el procesamiento de sustratos. Science 358:eaao0464. doi: 10.1126/science.aao0464
PubMed Abstract | CrossRef Full Text | Google Scholar
Ripstein, Z. A., Huang, R., Augustyniak, R., Kay, L. E., and Rubinstein, J. L. (2017). Estructura de una AAA+ unfoldase en el proceso de despliegue de sustrato. Elife 6:e25754. doi: 10.7554/eLife.25754
PubMed Abstract | CrossRef Full Text | Google Scholar
Snider, J., Thibault, G., y Houry, W. A. (2008). La superfamilia AAA+ de proteínas funcionalmente diversas. Genome Biol. 9:216. doi: 10.1186/gb-2008-9-4-216
PubMed Abstract | CrossRef Full Text | Google Scholar
Zehr, E., Szyk, A., Piszczek, G., Szczesna, E., Zuo, X., y Roll-Mecak, A. (2017). Las estructuras espirales y anulares de katanina arrojan luz sobre la carrera de potencia para el corte de microtúbulos. Nat. Struct. Mol. Biol. 24, 717-725. doi: 10.1038/nsmb.3448
PubMed Abstract | CrossRef Full Text | Google Scholar