Polimorfismo de un solo nucleótido
El polimorfismo de un solo nucleótido (SNP) se refiere a la diferencia de un solo nucleótido en la misma posición en la secuencia del ADN genómico. En general, una variación de un solo nucleótido con una frecuencia superior al 1% se denomina SNP. Los SNP implican sólo una variación de una base, que puede ser causada por una transición o transversión de una base. Hay aproximadamente un SNP por cada 1000 bases en el genoma humano, y el número total de SNPs en el genoma humano es de alrededor de 3 x 106.
SNP se ha convertido en la tercera generación de marcadores genéticos. Las características de los SNP los hacen más adecuados que otros marcadores polimórficos para los estudios de pleiotropía genética en rasgos y enfermedades complejas, y para el reconocimiento de genes basado en la población. Los SNP tienen una importancia significativa en la investigación biomédica. Los SNP se han utilizado en estudios de asociación de todo el genoma como marcadores de alta resolución en el mapeo de genes relacionados con enfermedades o rasgos normales. Los SNP sin un impacto observable en el fenotipo (denominados mutaciones silenciosas) también pueden ser útiles debido a su cantidad y a su herencia estable a lo largo de las generaciones.
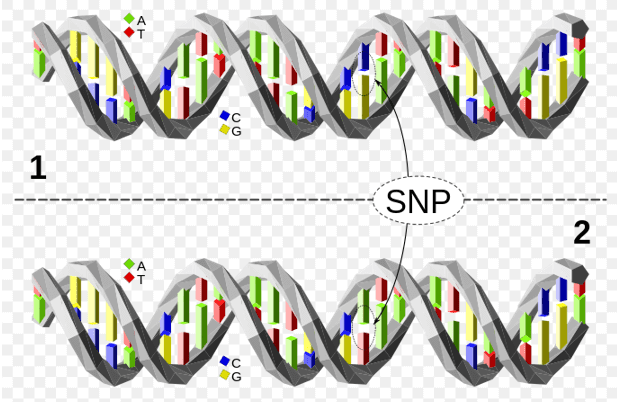 Figura 1. La molécula de ADN superior difiere de la molécula de ADN inferior en una ubicación de un solo par de bases (un polimorfismo C/A).
Figura 1. La molécula de ADN superior difiere de la molécula de ADN inferior en una ubicación de un solo par de bases (un polimorfismo C/A).
Microarray
El microarray de ADN se refiere a un chip de genes con un gran número de secuencias de ADN de sonda en una disposición específica inmovilizada en un sustrato sólido. El principio es la teoría de hibridación de ácidos nucleicos, y el ADN de la muestra detectada se hibrida con el microarray de ADN y se extiende. Posteriormente, los fragmentos de la reacción de unión no complementaria en el chip son lavados, y el chip de genes es sometido a un escaneo confocal por láser. A continuación, se miden las intensidades de las señales de fluorescencia y se interpretan como la abundancia de los diferentes genes a través de cierto procesamiento de datos.
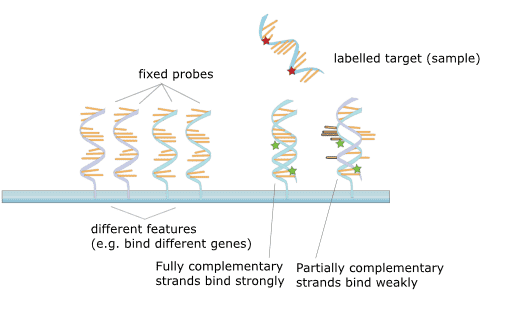 Figura 2. El principio del microarray.
Figura 2. El principio del microarray.
Microarray de SNP
El microarray de SNP utiliza secuencias de nucleótidos conocidas como sondas para hibridar con las secuencias de ADN analizadas, lo que permite el análisis cualitativo y cuantitativo de SNP mediante la detección de señales. En comparación con el método tradicional de diagnóstico unicelular, el microarray SNP es un método de alto rendimiento, que es capaz de realizar miles de reacciones en la superficie del chip de oligonucleótidos a la vez.
El flujo de trabajo del microarray SNP
El flujo de trabajo general del microarray SNP se muestra en la figura 3. En pocas palabras, hay varios procesos: Fabricación del chip SNP, preparación del ADN genómico de la muestra, hibridación y exploración por fluorescencia.
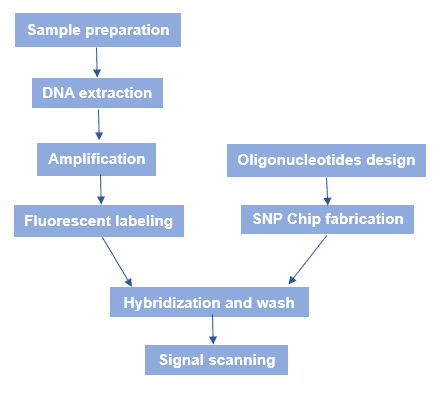 Figura 3. El flujo de trabajo de la micromatriz SNP.
Figura 3. El flujo de trabajo de la micromatriz SNP.
- Fabricación de chips SNP
Los chips de oligonucleótidos o ADNc prediseñados se disponen de forma ordenada y en alta densidad en el soporte de vidrio para hacer microarrays o chips de núcleo. Los métodos de fabricación del chip incluyen la síntesis in situ guiada por luz, el método de pulverización química, el método de recubrimiento de puntos por contacto, la síntesis controlada de ADN in situ, el método de impresión micromecánica sin contacto TOPSPOT® y la reproducción por litografía suave, etc. Ahora es posible colocar un total de 4×105 moléculas de ADN diferentes en un chip de 1 cm2. La tecnología GeneChip® de Affymetrix permite la síntesis de oligonucleótidos in situ de alta densidad, y ha sido pionera en este campo. - Preparación del ADN genómico de la muestra
En primer lugar, se prepara el material de la muestra. A continuación, se extrae el ADN genómico y se amplifica por PCR. Por último, se utilizan diferentes tintes fluorescentes para etiquetarlo. - Hibridación y escaneo
El ADN genómico etiquetado con fluorescencia se hibrida con el microarray de SNP en condiciones de reacción adecuadas. Después de ser lavado, se utiliza un escáner de fluorescencia específico para escanear la fluorescencia. A continuación, se realiza el análisis de imágenes por ordenador para procesar los datos antes de realizar el análisis bioinformático del gen diana.
En CD Genomics, nos dedicamos a proporcionar servicios fiables de genotipado SNP, incluyendo el genotipado por secuenciación, el microarray SNP, el genotipado SNP MassARRAY, el Hi-SNPseq, el sistema SNaPshot Multiplex para el genotipado SNP y el genotipado SNP TaqMan.
- Iwamoto K, Bundo M, Ueda J, et al. Detección de alteraciones estructurales cromosómicas en células individuales mediante arrays de SNP: un estudio sistemático del sesgo de amplificación y flujo de trabajo optimizado. Plos One, 2007, 2(12): e1306.
- Ho C C, Mun K S, Naidu R. Tecnología de matrices SNP: una matriz de esperanza en la investigación del cáncer de mama. Malaysian Journal of Pathology, 2013, 35(1):33-43.
- Ahmad A, Iqbal M A. Significance of genome-wide analysis of copy number alterations and UPD in myelodysplastic syndromes using combined CGH-SNP arrays. Current Medicinal Chemistry, 2012, 19(22).
- Sund K L, Zimmerman S L, Thomas C, et al. Regiones de homocigosidad identificadas por el análisis de microarrays SNP ayudan en el diagnóstico de enfermedades autosómicas recesivas y detectan incidentalmente las relaciones sanguíneas de los padres. Genetics in Medicine Official Journal of the American College of Medical Genetics, 2013, 15(1):70-78.
- Liu S, Zhou Z, Lu J, et al. Generación de SNPs asociados a genes a escala del genoma en el bagre para la construcción de un array de SNP de alta densidad. Bmc Genomics, 2011, 12(1):53.