Polymorphisme nucléotidique simple
Le polymorphisme nucléotidique simple (SNP) fait référence à la différence d’un seul nucléotide à la même position dans la séquence d’ADN génomique. En général, une variation d’un seul nucléotide dont la fréquence est supérieure à 1% est appelée un SNP. Les SNP n’impliquent qu’une seule variation de base, qui peut être causée par une seule transition ou transversion de base. Il y a environ un SNP pour 1000 bases dans le génome humain, et le nombre total de SNP dans le génome humain est d’environ 3 x 106.
Les SNP sont devenus la troisième génération de marqueurs génétiques. Les caractéristiques des SNP les rendent plus adaptés que d’autres marqueurs polymorphes aux études sur la pléiotropie génétique dans les traits et les maladies complexes, et à la reconnaissance des gènes dans les populations. Les SNP ont une importance significative dans la recherche biomédicale. Les SNP ont été utilisés dans des études d’association à l’échelle du génome comme marqueurs à haute résolution dans la cartographie des gènes liés aux maladies ou aux traits normaux. Les SNP sans impact observable sur le phénotype (appelés mutations silencieuses) peuvent également être utiles en raison de leur quantité et de leur héritage stable au fil des générations.
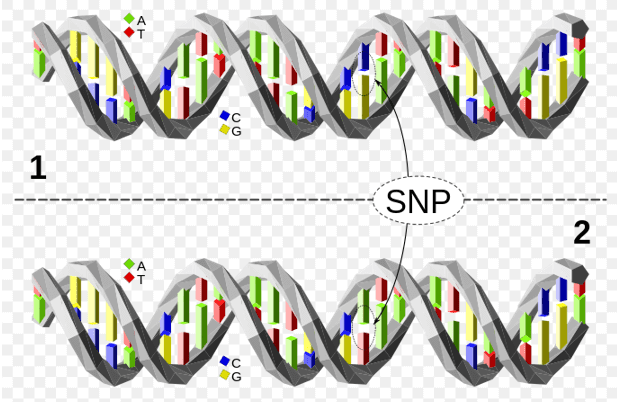 Figure 1. La molécule d’ADN supérieure diffère de la molécule d’ADN inférieure à un emplacement unique de paire de bases (un polymorphisme C/A).
Figure 1. La molécule d’ADN supérieure diffère de la molécule d’ADN inférieure à un emplacement unique de paire de bases (un polymorphisme C/A).
Microarray
Le microarray d’ADN se réfère à une puce génétique avec un grand nombre de séquences d’ADN sonde dans un arrangement spécifique immobilisé sur un substrat solide. Le principe est la théorie de l’hybridation des acides nucléiques, et l’ADN de l’échantillon détecté est hybridé avec la puce à ADN et étendu. Ensuite, les fragments de la réaction de liaison non complémentaire sur la puce sont lavés, et la puce génétique est soumise à un balayage confocal laser. Les intensités des signaux de fluorescence sont ensuite mesurées et interprétées comme l’abondance de différents gènes par le biais de certains traitements de données.
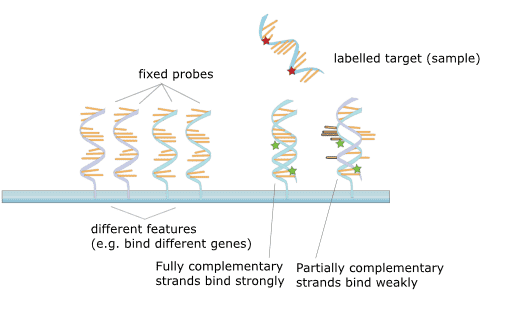 Figure 2. Le principe du microarray.
Figure 2. Le principe du microarray.
Microarray SNP
Le microarray SNP utilise des séquences nucléotidiques connues comme sondes pour s’hybrider avec les séquences d’ADN testées, permettant une analyse SNP qualitative et quantitative par la détection du signal. Par rapport à la méthode traditionnelle de diagnostic unicellulaire, la biopuce SNP est une méthode à haut débit, qui est capable d’effectuer des milliers de réactions sur la surface de la puce oligonucléotidique en une seule fois.
Le flux de travail de la biopuce SNP
Le flux de travail général de la biopuce SNP est présenté dans la figure 3. En bref, il y a plusieurs processus : La fabrication de la puce SNP, la préparation de l’ADN génomique de l’échantillon, l’hybridation et le balayage de fluorescence.
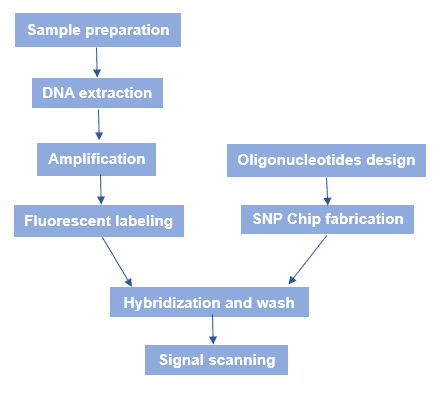 Figure 3. Le flux de travail de la micro-puce SNP.
Figure 3. Le flux de travail de la micro-puce SNP.
- Fabrication de la puce SNP
Les puces d’oligonucléotides ou d’ADNc préconçues sont disposées de manière ordonnée et en haute densité sur le support en verre pour faire des micro-puces ou des puces à noyau. Les méthodes de fabrication de la puce comprennent la synthèse in situ guidée par la lumière, la méthode de pulvérisation chimique, la méthode de revêtement par points de contact, la synthèse in situ contrôlée par l’ADN, la méthode d’impression micromécanique sans contact TOPSPOT® et la reproduction par lithographie douce, etc. Au total, 4 x 105 molécules d’ADN différentes peuvent désormais être placées sur une puce de 1 cm2. La technologie GeneChip® d’Affymetrix permet la synthèse in situ d’oligonucléotides à haute densité, et a été un pionnier dans ce domaine. - Préparation de l’ADN génomique de l’échantillon
D’abord, préparation du matériel de l’échantillon. Ensuite, l’ADN génomique est extrait et est amplifié par PCR. Enfin, différents colorants fluorescents sont utilisés pour le marquer. - Hybridation et balayage
L’ADN génomique marqué par fluorescence est hybridé avec la micro-puce SNP dans des conditions de réaction appropriées. Après avoir été lavé, un scanner de fluorescence spécifique est utilisé pour scanner la fluorescence. Ensuite, l’analyse d’image informatique est faite pour traiter les données avant l’analyse bioinformatique du gène cible.
À CD Genomics, nous nous consacrons à fournir des services de génotypage SNP fiables, y compris le génotypage par séquençage, la micro-puce SNP, le génotypage SNP MassARRAY, Hi-SNPseq, le système SNaPshot Multiplex pour le génotypage SNP et le génotypage SNP TaqMan.
- Iwamoto K, Bundo M, Ueda J, et al. Détection des altérations structurelles chromosomiques dans des cellules uniques par des tableaux SNP : une étude systématique du biais d’amplification et un flux de travail optimisé. Plos One, 2007, 2(12) : e1306.
- Ho C C, Mun K S, Naidu R. SNP array technology : an array of hope in breast cancer research. Malaysian Journal of Pathology, 2013, 35(1):33-43.
- Ahmad A, Iqbal M A. Significance of genome-wide analysis of copy number alterations and UPD in myelodysplastic syndromes using combined CGH-SNP arrays. Current Medicinal Chemistry, 2012, 19(22).
- Sund K L, Zimmerman S L, Thomas C, et al. Les régions d’homozygotie identifiées par l’analyse des microréseaux SNP aident à diagnostiquer une maladie autosomique récessive et à détecter incidemment les liens de parenté. Genetics in Medicine Official Journal of the American College of Medical Genetics, 2013, 15(1):70-78.
- Liu S, Zhou Z, Lu J, et al. Génération de SNP associés à l’échelle du génome chez le poisson-chat pour la construction d’un réseau SNP à haute densité. Bmc Genomics, 2011, 12(1):53.