Tutkimusaiheinen pääkirjoitus
AAA+ proteiinien rooli proteiinien korjaus- ja hajoamistoiminnassa. Protein Repair and Degradation
ATPases Associated with diverse cellular Activities (AAA+) comprise a superfamily of proteins that perform a large variety of functions essential to cell physiology, mukaan lukien proteiinien homeostaasin, DNA:n replikaation, rekombinaation, kromatiinin uudelleenmuotoilun, ribosomaalisen RNA:n prosessoinnin, molekulaarisen kohdentamisen, organellien biogeneesin ja membraanifuusion hallinta (Hanson ja Whiteheart, 2005; Erzberger ja Berger, 2006; Snider ym., 2008). Tämän superperheen jäsenille on ominaista niin sanottu AAA+-domeeni, joka sisältää ATP:n sitoutumiseen ja hydrolyysiin tarvittavat kanoniset Walker A- ja B-motiivit (Hanson ja Whiteheart, 2005). Tyypillisesti genomit koodaavat noin kymmenestä muutamaan sataan AAA+-perheen jäsentä (taulukko 1; Finn ym., 2017), joista jokaisen ajatellaan sopeutuneen tiettyihin toiminnallisiin kapeikkoihin, jotka edellyttävät täsmällisiä mekanismeja substraatin tunnistamiseen ja käsittelyyn (Hanson ja Whiteheart, 2005). AAA+-proteiinien silmiinpistävä adaptiivinen säteily toimia erilaisissa ympäristöissä havainnollistaa AAA+-domeenin monipuolista käyttökelpoisuutta (Erzberger ja Berger, 2006). AAA+-proteiinit muodostavat tyypillisesti heksaameerisia komplekseja ja toimivat moottoreina, jotka muokkaavat muita proteiineja, DNA/RNA:ta tai monikomponenttikomplekseja (kuva 1). Monet chaperonit ja ATP-riippuvaiset proteaasit ovatkin tai niillä on alayksiköitä, jotka kuuluvat tähän superperheeseen (kuva 1; Olivares ym., 2016).
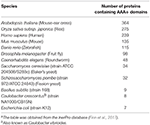
Taulukko 1. AAA-proteiinit. AAA+-proteiinien lukumäärä malliorganismeissaa.
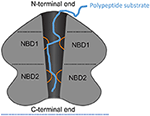
Kuva 1. Kaavio ClpA:sta esimerkkinä AAA+-heksameeristä. Kaavamainen esitys ClpA:n domeeniarkkitehtuurista ja vuorovaikutuksista esimerkkinä AAA+-heksaamerista, jossa on kaksi AAA+-domeenia per monomeeri. Tässä on esitetty ClpA-heksameerin sivukuva, joka kuvaa keskeistä, polypeptidiä johtavaa kanavaa. ClpA sisältää kolme domeenia, mukaan lukien N-terminaalinen domeeni ja kaksi AAA+-domeenia: nukleotideja sitovat domeenit 1 ja 2 (NBD1 ja NBD2). N-terminaalinen domeeni on vuorovaikutuksessa substraattispesifisyyden säätelijöiden kanssa, kun taas C-terminaalinen pää on vuorovaikutuksessa kammioproteaasi ClpP:n kanssa. NBD1:n ja NBD2:n liikkuvat silmukat (oranssi) työntyvät keskuskanavaan ja tarttuvat polypeptidisubstraattiin (sininen), mikä mahdollistaa ATP:n hydrolyysin kytkeytymisen polypeptidin translokaatioon keskuskanavan poikki.
Viime vuosina on tapahtunut huomattavaa edistystä useiden AAA+-proteiinien rakenteen ja toimintamekanismien selvittämisessä (Gates ym. ym., 2017; Puchades ym. ym. ym. ym. ym. ym. ym. ym. ym. ym. ym. ym. ym. ym. ym. ym. ym.). Tässä tutkimusaiheessa välitetään useita elementtejä tästä jännittävästä edistyksestä 21 artikkelissa, jotka käsittävät yksityiskohtaisen rakenteellisen ja mekanistisen näkemyksen useista AAA+-kapernaaseista ja proteaaseista, mm: ClpX (Alhuwaider ja Dougan; Bittner et al.; Elsholz et al.; LaBreck et al.; Vass et al.), ClpA (Bittner et al.; Duran et al.), ClpB ja Hsp104 (Chang et al.; Duran et al.; Franke et al.; Johnston et al.), Hsp78 (Abrahão et al.), ClpC (Alhuwaider ja Dougan; Elsholz et al.), ClpE (Elsholz et al.), Pontin (Mao ja Houry), Reptin (Mao ja Houry), FtsH (Alhuwaider ja Dougan), 19S-proteasomi (Snoberger et al.; Yedidi et al.), Lon (Alhuwaider ja Dougan; Bittner et al.; Fishovitz et al.), p97 (Hänzelmann ja Schindelin; Saffert ym.; Ye ym.), Pex1/6 (Saffert ym.), CbbQ (Mueller-Cajar), rubiscon aktivaasi (Bhat ym.), torsiinit (Chase ym.) ja mitokondriaaliset AAA+ proteaasit (Glynn). Tässä esittelemme näitä kiehtovia teoksia.
Tutkimuksia ClpXP:stä, Lonista ja niihin liittyvistä ATP-riippuvaisista proteaaseista
Tutkimusartikkelissaan ”The Protein Chaperone ClpX Targets Native and Non-native Aggregated Substrates for Remodeling, Disassembly, and Degradation with ClpP,” LaBreck et al. tekevät sarjan tyylikkäitä kokeita osoittaakseen, että ClpX:llä on disaggregaasiaktiivisuutta polypeptidejä kohtaan, jotka sisältävät erityisiä ClpX-tunnistussignaaleja (LaBreck ym.). ClpP:n läsnä ollessa ClpX kytkee näiden substraattien disaggregoitumisen niiden hajoamiseen. Tärkeää on myös todeta, että ClpXP estää ClpX-tunnistussignaaleja sisältävien proteiinien muodostamien aggregaattien kertymisen in vivo (LaBreck ym.). Nämä tutkimukset valaisevat ClpX:ää proteiinien disaggregaasina, joka oli aiemmin aliarvostettu.
Tutkimusartikkelissaan ”The Essential Role of ClpXP in Caulobacter crescentus Requires Species Constrained Substrate Specificity” (ClpXP:n olennainen rooli Caulobacter crescentus -kasvintuhoojissa vaatii lajikohtaista substraattispesifisyyttä) Vass et al. tutkivat ClpX:n lajispesifisiä toimintoja (Vass et al.). Kummallista kyllä, ClpX on välttämätön joissakin lajeissa, kuten C. crescentus, mutta ei välttämätön muissa bakteereissa, kuten E. coli (Vass ym.). Tärkeää on, että E. coli ClpX ei pystynyt täydentämään C. crescentus ClpX:ää in vivo (Vass ym.). Tämä aktiivisuuden puute johtui lajikohtaisista eroista ClpX:n N-terminaalisessa domeenissa, jotka ovat kriittisiä replikaatioklemmarin lataaja-alayksikön DnaX:n prosessoinnissa C. crescentuksessa. Näin ollen pienet erot ClpX:n spesifisyydessä voivat olla erityisen kriittisiä tietyille bakteerilajeille.
Katsauksessaan ”Functional Diversity of AAA+ Protease Complexes in Bacillus subtilis” Elsholz et al. käsittelevät useiden AAA+-proteaasien tehtäviä B. subtiliksessa, nimittäin: ClpCP, ClpEP, ClpXP, ClpYQ, LonA/B ja FtsH (Elsholz ym.). He käsittelevät sitä, miten erilaiset stressivasteet säätelevät niiden ilmentymistä ja fenotyyppejä, joita havaitaan näiden eri proteaasien poistamisen yhteydessä. Joidenkin näistä proteaaseista kykyä kontrolloida kompetenssia, sporulaatiota, motiliteettia ja biofilmin muodostumista kuvataan. Lopuksi kirjoittajat keskustelevat näiden proteaasien kohdentamisesta uusien antibioottien kehittämiseksi.
Katsauksessaan ”AAA+ Machines of Protein Destruction in Mycobacteria” (AAA+ proteases of Protein Destruction in Mycobacteria) Alhuwaider ym. (Alhuwaider ja Dougan) käsittelevät viimeaikaisia edistysaskeleita, jotka ovat tapahtuneet mykobakteerien AAA+-proteaasien rakenteen ja toiminnan määrittämisessä. Nämä proteaasit ovat: ClpXP1P2, ClpC1P1P2, Lon, FtsH ja Mpa. Kirjoittajat käsittelevät myös mykobakteereissa esiintyvää Pup-proteasomijärjestelmää (PPS), joka vastaa eukaryoottien ubikitiini-proteasomijärjestelmää. Alhuwaider et al. päättävät artikkelinsa keskusteluun uusista yhdisteistä, jotka häiritsevät tai estävät ClpP1P2:n toimintaa, ja muista yhdisteistä, jotka häiritsevät ClpC1:n toimintaa. Näillä yhdisteillä on lupaavia vaikutuksia mykobakteereja vastaan.
Tutkimusartikkelissa ”The Copper Efflux Regulator CueR Is Subject to ATP-Dependent Proteolysis in Escherichia coli” (Kupari-evluksisäätäjä CueR altistuu ATP-riippuvaiselle proteolyysille Escherichia coli -bakteerissa) Bittner et al. osoittavat, että AAA+-proteaasit Lon, ClpXP ja ClpAP ovat vastuussa kupari-evluksisäätelyjärjestelmän (Copper Efflux Regulator CueR system) induktiota kontrolloivien transkriptionaalisten faktorien, CueR:n (Bittner et al.), hajoamisesta. Kirjoittajat havaitsivat, että AAA+-proteaasien CueR:n tunnistaminen edellyttää CueR:n saavutettavaa C-terminaalia. He päättelevät, että ATP-riippuvaiset proteaasit ovat välttämättömiä kuparin homeostaasille E. colissa.
Fishovitz ym. suorittavat yksityiskohtaisen vertailun ihmisen ja E. colin Lon välillä tutkimusartikkelissaan ”Utilization of Mechanistic Enzymology to Evaluate the Significance of ADP Binding to Human Lon Protease” (Fishovitz ym.). Yksityiskohtaisen mekanistisen tutkimuksen avulla he havaitsivat, että toisin kuin E. coli Lonilla, ihmisen Lonilla on alhainen affiniteetti ADP:hen huolimatta siitä, että ATPaasiaktiivisuuden kcat- ja KM-arvot ovat vertailukelpoisia. He ehdottavat, että ihmisen Lonia ei säädellä substraatin edistämällä ADP/ATP-vaihtomekanismilla. Nämä erot ihmisen ja E. coli Lonin välillä saattavat mahdollistaa lajispesifisten Lon-inhibiittorien kehittämisen tulevaisuudessa.
Tohtori Glynn käsittelee katsauksessaan ”Multifunctional Mitochondrial AAA Proteases” kahta mitokondriaalista AAA-proteaasia, i-AAA:ta ja m-AAA:ta (Glynn). Molemmat ovat mitokondrioiden sisemmän kalvon proteiineja. Kuitenkin i-AAA projisoi ATPaasi- ja proteaasidomeenit mitokondrioiden väliseen kalvotilaan, kun taas m-AAA-proteaasi projisoi katalyyttiset domeenit matriisiin. Näiden proteaasien rakenteita ja niiden toimintamekanismeja käsitellään. Proteaasit voivat suorittaa täydellisen substraatin hajoamisen, mutta ne voivat myös pilkkoa vain tiettyjä substraatteja, kuten MrpL32:n ja Atg32:n osalta.
ClpB ja Hsp104
Mini-katsauksessaan ”Structural Elements Regulating AAA+ Protein Quality Control Machines” Chang ym. keskustelevat siitä, miten huokossilmukka-1, Inter-Subunit Signaling -motiivi ja Pre-Sensor I -insertti-motiivi saattavat vaikuttaa kahden Hsp100-disaggregaasin, bakteerien ClpB:n ja hiivan Hsp104:n, toimintaan (Chang ym.). He ehdottavat mallia siitä, miten nämä rakenneosat voisivat mahdollistaa AAA+-ATPaasi-syklin kytkemisen substraatin translokaatioon ClpB:n ja Hsp104:n keskuskanavan läpi. He käsittelevät näiden kolmen AAA+ -proteiinin (ClpA, ClpB ja Hsp104) kykyä translokoida polypeptidejä heksameeristen kompleksiensa kautta. Kaikilla näillä proteiineilla on kaksi AAA+-domeenia, ja niiden tiedetään purkavan proteiineja. Tärkeää on, että ClpB:n ja Hsp104:n tiedetään toimivan myös disaggregaaseina, kun taas ClpA voi muodostaa kompleksin ClpP-proteaasin kanssa. Kirjoittajat korostavat tarvetta käyttää transienttitilakineettisiä menetelmiä näiden moottoriproteiinien kineettisten mekanismien tutkimiseksi. He kuvaavat, miten tällaisten menetelmien avulla he pystyivät osoittamaan, että esimerkiksi ClpA translokoi polypeptidejä noin 20 aa s-1:n nopeudella, kun taas ClpP-proteaasin kanssa kompleksissa ClpA:n translokaationopeus on vieläkin suurempi, noin 35 aa s-1. Kirjoittajat käsittelevät myös Hsp70-kapersonin merkitystä ClpB/Hsp104:n toiminnassa ja havaintoa lajispesifisyydestä Hsp70:n ja ClpB/Hsp104:n välisessä vuorovaikutuksessa.
Tutkimusartikkelissaan ”Mutant Analysis Reveals Allosteric Regulation of ClpB Disaggregase” (Mutanttianalyysi paljastaa ClpB-disaggregaasin allosterisen sääntelyn), Franke et al. toteuttavat mutaatioanalyysin E. colin ClpB-disaggregaasille luonnehtimalla sen allosterista säätelyä (”Mutant Analysis Reveals Allosteric Regulation of ClpB Disaggregase of ClpB Disaggregase”), (Franke et al.). ClpB voidaan jakaa N-terminaaliseen domeeniin ja kahteen AAA+-domeeniin, jotka erottaa toisistaan M-domeeniksi kutsuttu kierteinen alue. Kirjoittajat tunnistavat ensimmäisessä AAA+-domeenissa erittäin konservoituneen jäännöksen, A328. ClpB-A328V-mutantilla todettiin olevan erittäin korkea ATPaasiaktiivisuus ja se oli solutoksinen. Odottamatta ClpB-A328V:n korkea ATPaasiaktiivisuus johtui pääasiassa toisesta AAA+-renkaasta, kuten arvioitiin amidivetyvaihdon massaspektrometrialla. Kirjoittajat päättelevät, että A328 on ratkaiseva residuaali ClpB:n molempien AAA+-renkaiden ATP-hydrolyysin kontrolloinnissa.
Tutkimusartikkelissaan ”Substrate Discrimination by ClpB and Hsp104” Johnston et al. kuvaavat ClpB:n ja Hsp104:n synnynnäisiä substraattipreferenssejä DnaK- ja Hsp70-kapernaattisysteemien poissaollessa (”Substrate Discrimination by ClpB and Hsp104 by ClpB and Hsp104 and Hsp104 and Hsp104 and Substrate Discrimination by ClpB and Hsp104 by Substrate Discrimination by ClpB and Hsp104 and Substrate Discrimination”). He osoittavat, että substraattispesifisyys määräytyy kunkin proteiinin ensimmäisen AAA+-domeenin mukaan. He pääsivät tähän johtopäätökseen testaamalla näiden kahden chaperonin kykyä toimia useisiin mallisubstraatteihin. He testasivat myös kahden chaperonin erilaisia kimeeroja.
Kirjassa ”Hsp78 (78 kDa Heat Shock Protein), a Representative AAA Family Member Found in the Mitochondrial Matrix of Saccharomyces cerevisiae” Abrahão et al. käsittelevät Hsp78:n rakennetta ja toimintaa (Abrahao et al.). Hsp78 on mitokondriaalinen paralogi Hsp104:lle, joka toimii proteiinien hajoamisessa ja uudelleenaktivoinnissa (Abrahao et al.). Kummallista kyllä, Hsp104 ja Hsp78 katosivat siirryttäessä alkueläimistä metazoiksi (Abrahao ym.). Abrahao et al. keskustelevat kuitenkin ANKCLP:n olemassaolosta, joka esiintyy Hsp78:n ja Hsp104:n rinnalla alkueläimissä ja selviytyy evolutiivisesta siirtymisestä metazooihin. ANKCLP:llä on AAA+-domeeni, joka muistuttaa Hsp104:n ja Hsp78:n nukleotideja sitovaa domeenia 2 (NBD2), mutta on muuten hyvin erilainen. Mielenkiintoista on, että ANKCLP:n mutaatiot aiheuttavat ihmisillä 3-metyyliglutakonihappouriaa, etenevää aivojen surkastumista, älyllistä kehitysvammaisuutta, synnynnäistä neutropeniaa, harmaakaihia ja liikehäiriöitä (Abrahao ym.).
p97
Katsauksessaan ”Structure and Function of p97 and Pex1/6 Type II AAA+ Complexes” Saffert ym. käsittelevät kahta erilaista AAA+-kompleksia, jotka uudelleenmuotoilevat ubikitoituneita substraattiproteiineja (Saffert ym.). Yksi p97:n tehtävä on siirtää ubikitinoidut substraatit ER-kalvolta proteasomiin ER-assosioituneen hajoamisen aikana (Saffert ym.). Pex1/Pex6 on sitä vastoin heteroheksameerinen moottori, joka koostuu vuorotellen Pex1- ja Pex6-alayksiköistä ja joka on välttämätön peroksisomin biogeneesille ja toiminnalle. P97:n ja Pex1/6:n viimeaikaisia kryoelektronimikroskooppisia (cryo-EM) rakenteita käsitellään ja keskeisiä rakenteellisia eroja korostetaan.
Katsauksessaan ”A Mighty ’Protein Extractor’ of the Cell: Structure and Function of the p97/CDC48 ATPase” Ye ym. tiivistävät nykytietämyksensä p97:n rakenteesta ja toiminnasta sekä sen roolista useissa taudeissa (Ye ym.). p97:llä on kaksi lyhyen linkkeriosuuden yhdistämää AAA+- domeenia. Sillä on myös N-terminaalinen domeeni, joka välittää sen vuorovaikutusta eri adaptoriproteiinien kanssa. Kirjoittajat käsittelevät yksityiskohtaisesti p97:n rakennetta ja nukleotidien vaikutusta sen eri konformaatioihin. Tutkimukset perustuvat EM:n, röntgenkristallografian ja nopean atomivoimamikroskopian kaltaisten tekniikoiden käyttöön. Tämän jälkeen kirjoittajat käsittelevät tämän erittäin konservoituneen proteiinin monisoluisia toimintoja, mukaan lukien sen roolit ER-assosioituneessa proteiinien hajoamisessa (ERAD), mitokondrioihin assosioituneessa hajoamisessa (MAD) irrottamalla polypeptidejä mitokondrioiden ulkokalvolta ja ribosomeihin assosioituneessa hajoamisessa (RAD). Lopuksi Ye et al. esittävät yhteenvedon p97-mutaatioista, jotka johtavat useisiin ihmisillä esiintyviin sairauksiin, kuten IBMPFD (Inclusion Body Myopathy associated with Paget’s disease of the bone and Frontotemporal Dementia)], FALS (familiaalinen amyotrofinen lateraaliskleroosi), CMT2Y (Charcot-Marie-Toothin tauti, tyyppi 2Y), perinnöllinen spastinen paraplegiasairaus (Hereditary Spastic Paraplegias, HSP), Parkinsonin tauti (Parkinson’s Disease, Parkinson’s Disease) ja Alzheimerin tauti (AD).
Hänzelmann ja Schindelin käsittelevät p97:ää käsittelevässä katsauksessaan ”The Interplay of Cofactor Interactions and Post-translational Modifications in the Regulation of the AAA+ ATPase p97” (Kofaktori-interaktioiden ja post-translationaalisten modifikaatioiden vuorovaikutus AAA+-ATPaasi p97:n säätelyssä) sitä, miten erilaiset kofaktorit muokkaavat p97-ATPaasin aktiivisuutta (Hänzelmann ja Schindelin). He korostavat, että p97:n kyky osallistua lukuisiin soluprosesseihin johtuu tämän proteiinin kanssa vuorovaikutuksessa olevien kofaktorien suuresta määrästä. Ne selvittävät kolme erilaista p97-kofaktoreiden luokkaa, nimittäin: (UBA-UBX-proteiinien ja UFD1-NPL4:n kaltaiset substraattia rekrytoivat kofaktorit, ii) substraattia käsittelevät kofaktorit, kuten ubikitiini- (E3-) ligaasit ja deubikitinaasit (DUB:t), ja iii) UBX-proteiinien kaltaiset säätelyyn liittyvät kofaktorit, jotka voivat sekventoida tai kierrättää p97-heksaamereita. Kirjoittajat käsittelevät myös posttranslationaalisten modifikaatioiden merkitystä p97:n aktiivisuuteen sekä sen vuorovaikutukseen kofaktoriensa ja substraattiensa kanssa.
AAA+ Proteins of the Proteasome
In ”AAA-ATPases in Protein Degradation”, Yedidi et al. tarkastelevat eukaryoottisen proteasomin AAA+-ATPaasien Rpt1, Rpt2, Rpt3, Rpt4, Rpt5 ja Rpt6 sekä joidenkin niiden bakteerisukulaisten, kuten PAN:n, Mpa:n ja VAT:n toimintaa (Yedidi ym.). He keskittyvät uusiin teknologioihin ymmärtääkseen, miten nämä AAA+-ATPaasit toimivat translokoimalla taitamattomia polypeptidejä proteaasin proteolyyttiseen kammioon (Yedidi et al.). Konformaatiomuutokset AAA+ -renkaan ja viereisen proteaasikammion sisällä näyttävät synnyttävän peristalttisen pumppausmekanismin, joka kuljettaa substraatteja hajotettavaksi (Yedidi et al.).
Tutkimusartikkelissaan ”Proteasomaaliset ATP-taasit käyttävät hidasta, mutta erittäin proseduraalista strategiaa proteiinien avaamiseen” Snoberger et al. toteavat, että proteasomaaliset AAA+-proteiinit käyttävät hidasta, mutta erittäin prosessiivista moottorimekanismia toimittaakseen substraatteja proteasomin proteolyyttiseen onteloon (Snoberger ym.). Tämä mekanismi on ristiriidassa ClpX:n kanssa, joka käyttää suuren nopeuden mutta vähemmän prosessoivaa moottorimekanismia toimittaakseen substraatteja ClpP-proteaasille hajotettavaksi. Nämä motoristen mekanismien erot ovat saattaneet kehittyä vastauksena niiden spesifisen asiakaskunnan erilaisiin vaatimuksiin.
Rubisco-aktivaasit
Katsauksessaan ”Rubisco Activases: AAA+ Chaperones Adapted to Enzyme Repair” Bhat et al. käsittelevät Rubisco-aktivaasin (Rca) ainutlaatuista tehtävää Rubiscon uudelleenmuotoilussa (Bhat et al.). Rca on AAA+-chaperoni, joka on hyvin konservoitunut fotosynteettisissä organismeissa bakteereista korkeampiin kasveihin. Rubisco on ribuloosi-1,5-bisfosfaattikarboksylaasi/oksygenaasientsyymi, joka osallistuu ilmakehän hiilidioksidin sitomiseen fotosynteesin aikana. Se on maapallon runsain proteiini ja avainentsyymi planeetan kaiken orgaanisen aineen synteesissä. Rubisco on kuitenkin huono entsyymi, ja sen katalyyttisten reaktioiden sivutuotteet tai joidenkin kasvien matalissa valo-olosuhteissa syntetisoimat yhdisteet estävät sitä helposti. Rca:n tehtävänä on lievittää tai ”parantaa” Rubisco tällaisista ongelmallisista estoista. Kirjoittajat käsittelevät eri lajeista peräisin olevan Rca:n rakennetta ja sen mahdollisia toimintamekanismeja.
Tohtori Mueller-Cajar esittää katsauksen aiheesta ”The Diverse AAA+ Machines that Repair Inhibited Rubisco Active Sites” (Mueller-Cajar). Hän käsittelee kolmea evolutiivisesti erilaista Rubisco-aktivaasien (Rcas) luokkaa: (1) vihreät ja (2) punaiset Rcas-tyypit, joita esiintyy lähinnä vihreän ja punaisen plastidilinjan fotosynteettisissä eukaryooteissa, ja (3) kemoautotrofisissa bakteereissa esiintyvä CbbQO. Hän käsittelee näiden aktivaasien evoluutiota ja niiden mahdollista käyttöä synteettisessä biologiassa Rubiscon aktiivisuuden lisäämiseksi kasveissa.
Torsin
Heidän näkökulma-artikkelissaan ”Torsin ATPases: Harnessing Dynamic Instability for Function” Chase et al. käsittelevät torsineja, jotka ovat myös fylogeneettisesti sukua hiivan Hsp104:n NBD2:lle (Chase et al.). Torsiinit ovat ainoat AAA+-ATPaasit, jotka ovat paikallistuneet ER:n ja siihen kytkeytyneen ydinkuoren sisälle (Chase et al.). Mielenkiintoista on, että TorsinA:n mutaatiot aiheuttavat DYT1-dystoniaa, neurologista häiriötä ihmisillä (Chase ym.). Torsineilla on heikko ATPaasiaktiivisuus, jota lisätään aktiivisen alueen komplementaatiolla, joka johtuu yhteiskokoonpanosta spesifisten apukofaktoreiden LAP1 ja LULL1 kanssa (Chase ym.). Chase ym. viittaavat siihen, että Torsinin ja kofaktorikompleksien dynaamisella kokoonpanolla ja purkamisella on tärkeä rooli niiden toiminnassa ydinliikenteessä ja ydin- ja huokoskompleksien kokoonpanossa (Chase ym.).
Pontin ja Reptin
Mao ja Houry käsittelevät laajassa katsauksessaan ”The Role of Pontin and Reptin in Cellular Physiology and Cancer Etiology” (Pontinin ja Reptinin rooli solufysiologiassa ja syövän etiologiassa) pitkälle konservoituneiden Pontinin ja Reptinin AAA+-ATPaasien moninaisia toimintoja (Mao ja Houry). Nämä kaksi proteiinia toimivat tyypillisesti yhdessä kompleksina, mutta voivat toimia myös itsenäisesti. Kirjoittajat korostavat Pontinin ja Reptinin roolia kromatiinin uudelleenmuokkauksessa. He käsittelevät myös sitä, miten Pontin ja Reptin moduloivat useiden proto-onkogeenien, kuten MYC:n ja β-kateniinin, transkriptiotoimintaa. Mao ja Houry selvittävät, miten Pontinin ja Reptinin on havaittu olevan välttämättömiä PIKK-signalointikompleksien sekä telomeraasin, mitoottisen karan, RNA-polymeraasi II:n ja snoRNP:iden kokoamisessa. Kirjoittajat esittävät lopuksi katsauksen nykyisistä ponnisteluista, joilla pyritään tunnistamaan Pontinin ja Reptinin estäjiä, joita voitaisiin kehittää uusina syöpien vastaisina aineina.
Loppuhuomautukset
Johtopäätöksenä voidaan todeta, että tässä 21 artikkelista koostuvassa artikkelikokoelmassa tuodaan esiin useita tärkeitä rakenteellisia ja mekanistisia näkökohtia, jotka koskevat proteiinien korjaukseen ja hajoamiseen osallistuvia AAA+-proteiineja. Odotamme innolla, miten ala kehittyy edelleen meneillään olevan kryo-EM-vallankumouksen aikana (Egelman, 2016). Odotamme, että kryo-EM mahdollistaa syvemmän ymmärryksen siitä, miten nämä kiehtovat molekyylikoneet toimivat erilaisissa tilanteissa (Gates ym., 2017; Puchades ym., 2017; Ripstein ym., 2017; Zehr ym, 2017).
Tekijöiden panos
Kaikki luetellut kirjoittajat ovat antaneet merkittävän, suoran ja älyllisen panoksen työhön ja hyväksyneet sen julkaistavaksi.
Osaajien eturistiriitoja koskeva lausunto
Tekijät ilmoittavat, että tutkimus suoritettiin ilman kaupallisia tai taloudellisia suhteita, jotka voitaisiin tulkita mahdolliseksi eturistiriidaksi.
Kiitokset
Työ WH:n laboratoriossa tällä alalla on rahoitettu Kanadan terveystutkimuslaitosten projektitutkimusavustuksella (PJT-148564). JS:n työtä tukee NIH:n apuraha R01GM099836.
Egelman, E. H. (2016). Kryo-EM:n nykyinen vallankumous. Biophys. J. 110, 1008-1012. doi: 10.1016/j.bpj.2016.02.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Erzberger, J. P., and Berger, J. M. (2006). AAA+-proteiinien evolutiiviset suhteet ja rakenteelliset mekanismit. Annu. Rev. Biophys. Biomol. Struct. 35, 93-114. doi: 10.1146/annurev.biophys.35.040405.101933
PubMed Abstract | CrossRef Full Text | Google Scholar
Finn, R. D., Attwood, T. K., Babbitt, P. C., Bateman, A., Bateman, A., A., Bork, P., Bridge, A. J., ym. et al. (2017). InterPro in 2017-beyond protein family and domain annotations. Nucleic Acids Res. 45, D190-D199. doi: 10.1093/nar/gkw1107
PubMed Abstract | CrossRef Full Text | Google Scholar
Gates, S. N., Yokom, A. L., Lin, J., Jackrel, M. E., Rizo, A. N., Kendsersky, N. M., et al. (2017). AAA+-disaggregaasi Hsp104:n AAA+:n kaltaisen polypeptidin translokaatiomekanismi. Science 357, 273-279. doi: 10.1126/science.aan1052
PubMed Abstract | CrossRef Full Text | Google Scholar
Hanson, P. I., and Whiteheart, S. W. (2005). AAA+-proteiinit: on moottori, toimii. Nat. Rev. Mol.Cell Biol. 6, 519-529. doi: 10.1038/nrm1684
PubMed Abstract | CrossRef Full Text | Google Scholar
Olivares, A. O., Baker, T. A., and Sauer, R. T. (2016). Mekanistisia oivalluksia bakteerien AAA+-proteaaseista ja proteiinien uudelleenmuokkauskoneista. Nat. Rev. Microbiol. 14, 33-44. doi: 10.1038/nrmicro.2015.4
PubMed Abstract | CrossRef Full Text | Google Scholar
Puchades, C., Rampello, A. J., Shin, M., Giuliano, C. J., Wiseman, R. L., Glynn, S. E., et al. (2017). Mitokondrioiden sisemmän kalvon AAA+-proteaasi YME1:n rakenne antaa tietoa substraatin käsittelystä. Science 358:eaao0464. doi: 10.1126/science.aao0464
PubMed Abstract | CrossRef Full Text | Google Scholar
Ripstein, Z. A., Huang, R., Augustyniak, R., Kay, L. E., ja Rubinstein, J. L. (2017). AAA+ unfoldaasin rakenne substraatin purkamisprosessissa. Elife 6:e25754. doi: 10.7554/eLife.25754
PubMed Abstract | CrossRef Full Text | Google Scholar
Snider, J., Thibault, G., ja Houry, W. A. (2008). Toiminnallisesti erilaisten proteiinien AAA+-superperhe. Genome Biol. 9:216. doi: 10.1186/gb-2008-9-4-216
PubMed Abstract | CrossRef Full Text | Google Scholar
Zehr, E., Szyk, A., Piszczek, G., Szczesna, E., Zuo, X., ja Roll-Mecak, A. (2017). Kataniinin spiraali- ja rengasrakenteet valaisevat mikrotubulusten katkaisun voimansiirtoa. Nat. Struct. Mol. Biol. 24, 717-725. doi: 10.1038/nsmb.3448
PubMed Abstract | CrossRef Full Text | Google Scholar