Data la relativa semplicità strutturale e la ripetitività del DNA, ne consegue che le proteine che si legano specificamente ad esso potrebbero avere motivi comuni del dominio legante il DNA ma con catene laterali di aminoacidi specifici che permettono interazioni di legame specifiche.
- elica-gira-elica: trovata nelle proteine procariotiche di legame al DNA.
Figura: elica-gira-elica
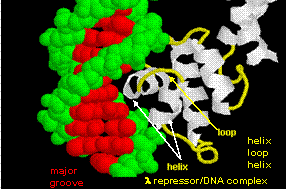
Le figure mostrano due proteine di questo tipo, il repressore cro del batteriofago 434 e il repressore lambda del batteriofago lambda. (I batteriofagi sono virus che infettano i batteri.) Notate come la specificità è ottenuta, in parte, dalla formazione di specifici legami H tra la proteina e il solco maggiore del DNA operatore.
Figura: Complesso Lambda Repressore/DNA
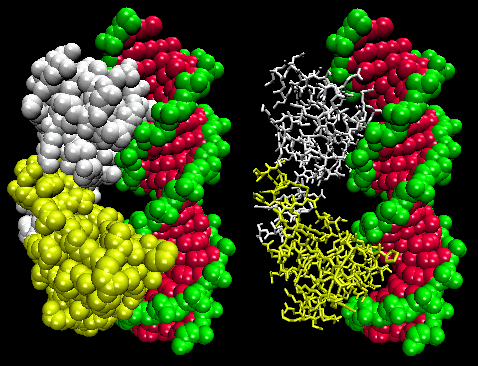
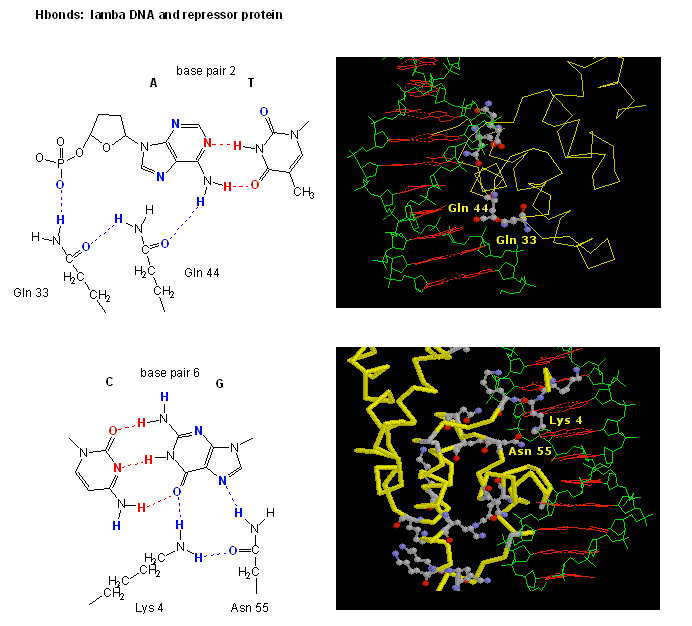
Figura: Interazioni H Bond tra il repressoreλ e il DNA
![]() Jmol: Aggiornato Lambda Repressor/DNA complex Jmol14 (Java) | JSMol (HTML5)
Jmol: Aggiornato Lambda Repressor/DNA complex Jmol14 (Java) | JSMol (HTML5)
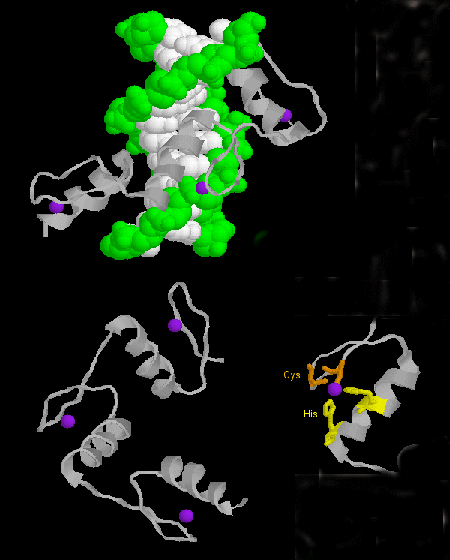
- zinc finger: (eucarioti) Queste proteine hanno un motivo di sequenza comune di X3-Cys-X2-4-Cys-X12-His-X3-4-His-X4- in cui X è qualsiasi amminoacido. Lo Zn2+ è coordinato tetraedricamente con le catene laterali Cys e His, che si trovano rispettivamente su uno dei due filamenti beta antiparalleli e su un’elica alfa. Il dito di zinco, stabilizzato dallo zinco, si lega al solco maggiore del DNA. ]
Figura: dito di zinco
![]() Jmol: Aggiornato Zif268:Complesso DNA Jmol14 (Java) | JSMol (HTML5)
Jmol: Aggiornato Zif268:Complesso DNA Jmol14 (Java) | JSMol (HTML5)
Le proteine Zn finger, di cui 900 sono codificate nel genoma umano (compresa la proteina legante gli isolanti eucariotici CTCF descritta sopra) possono essere mobilitate per riparare effettivamente specifiche mutazioni nelle cellule, che se effettuate in una percentuale abbastanza alta di cellule mutanti potrebbero curare specifiche malattie genetiche come alcune forme di immunodeficienza combinata grave. In questa nuova tecnica (Urnov et al, 2005), più domini di legame Zn finger collegati, (uno di quelli naturali o forme mutanti prodotte in laboratorio), ognuno specifico per una certa sequenza nucleotidica, è collegato ad una endonucleasi non specifica, derivata dall’enzima FokI. La nucleasi è attiva in forma dimerica quindi il complesso attivo richiede due domini endonucleasi, ciascuno legato a quattro diversi domini Zn finger, per assemblarsi al sito bersaglio. La specificità del legame è ottenuta mediante selezione da parte dei domini Zn finger. Una intaccatura è quindi fatta dal DNA dalla nucleasi, e i meccanismi di riparazione della cellula ospite seguono. Questo processo comporta la separazione dei filamenti, la ricombinazione omologa della regione intaccata con il DNA complementare all’interno della cellula e la riparazione dell’intaccatura. Se un eccesso di DNA wild type (non mutato) viene aggiunto alle cellule e usato come modello, la normale mutazione di riparazione del DNA fisserebbe la mutazione. Urnov et al hanno dimostrato che fino al 20% delle cellule coltivate contenenti una mutazione possono essere riparate in laboratorio. Se queste cellule ottengono un vantaggio selettivo di crescita, le cellule mutate verrebbero alla fine sostituite da cellule di tipo selvaggio.
- recettori degli ormoni steroidei: (eucarioti) In contrasto con la maggior parte degli ormoni, che si legano ai recettori della superficie cellulare, gli ormoni steroidei (derivati del colesterolo) passano attraverso la membrana cellulare e si legano ai recettori citoplasmatici attraverso un dominio di legame ormonale. Questo cambia la forma del recettore che poi si lega a un sito specifico sul DNA (elemento di risposta ormonale) attraverso un dominio di legame al DNA. In una struttura analoga a quella dello zinc finger, lo Zn 2+ è coordinato tetraedricamente a 4 Cys, in una struttura di tipo globulare che si lega come dimero a due sequenze identiche ma invertite di DNA (palindromo) all’interno del solco maggiore. (Esempi di palindromi: Able was I before I saw Elba; Dennis and Edna dine, said I, as Enid and Edna sinned.
Considerate il recettore dei glucocorticoidi (GR) come esempio specifico. Si lega al DNA come un dimero. I due domini di legame al DNA del dimero si associano a due scanalature principali adiacenti del DNA nella sequenza di legame del GR (GBS), una breve sequenza di DNA all’interno del promotore. Meijsing, et al. hanno scoperto che non solo la GBS agisce come un sito di legame per la GR, permettendo la trascrizione dei geni, ma influenza anche la conformazione del recettore, causando la trascrizione dei geni da regolare in un altro modo. Il gruppo ha costruito dei “geni reporter” di luciferasi che hanno GBS collegato al gene per la proteina luciferasi, che esprimerebbe la proteina luciferasi (che diventa fluorescente) se fossero trascritti, con la GBS. Hanno scoperto che l’attività trascrizionale relativa non era correlata all’affinità di legame relativo del GR al GBS. I GBS che erano molto più attivi di altri si legavano in modo comparabile con quelli di minore attività, mentre i GBS con attività trascrizionale simile si legavano con affinità diverse. Questo dimostra che la GBS sta conferendo una funzione unica alla GR ad essa associata (cioè la trascrizione non è semplicemente influenzata dal fatto che la GR sia legata o meno alla GBS). Si è scoperto che un “braccio di leva” del recettore subisce cambiamenti conformazionali quando è legato al DNA, con cambiamenti specifici alla sequenza a cui è legato. Una proteina mutante, GR-γ, è stata resa identica alla proteina wild-type, GR-α, eccetto che per il braccio di leva è stata trovata ad avere un’attività trascrizionale diversa anche se si legavano allo stesso sito sul DNA, dimostrando che il braccio di leva e la sua conformazione influenzano la trascrizione.
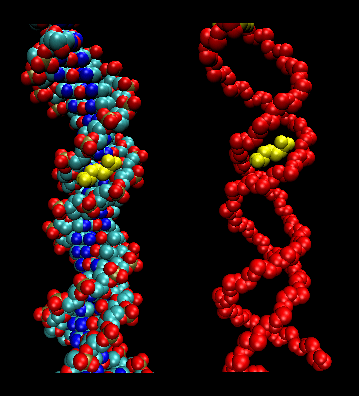
- leucina zip (o forbici): (eucarioti) Queste proteine contengono tratti di 35 aminoacidi in cui Leu si trova ripetutamente a intervalli di 7 aminoacidi. Queste regioni della proteina formano eliche anfifile, con Leu su una faccia, un Leu dopo due giri di elica. Due di queste proteine possono formare un dimero, stabilizzato dal legame di queste eliche anfifiliche non polari e ricche di leucina l’una con l’altra, formando una spirale, proprio come nella proteina muscolare miosina. La cerniera di leucina rappresenta il dominio di legame della proteina. Il dominio di legame al DNA si trova nei primi 30 amminoacidi N-terminali, che sono basici e formano un’alfa-elica quando la proteina si lega al DNA. La cerniera di leucina funziona quindi per unire due proteine leganti il DNA, permettendo alle eliche di basi N-terminali di interagire con il solco maggiore del DNA in modo specifico per la base. Valina e isoleucina, insieme alla leucina, si trovano spesso in tratti di aminoacidi che possono interagire per formare altri tipi di spirali.
Figura: leucina zipper (realizzata con VMD)
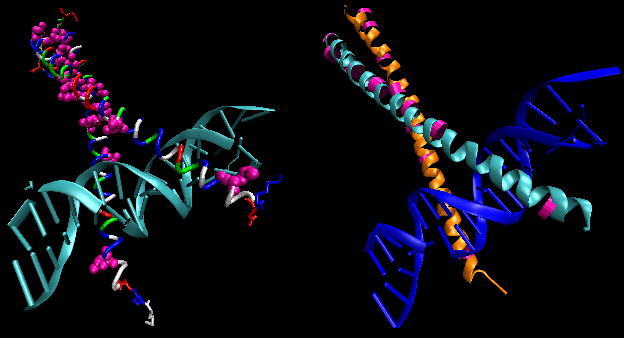
![]() Jmol: Aggiornato Leucine Zipper Jmol14 (Java) | JSMol (HTML5)
Jmol: Aggiornato Leucine Zipper Jmol14 (Java) | JSMol (HTML5)
Così come le nucleasi Zinc fingers sono state utilizzate per indurre la riparazione delle mutazioni, un altro studio sul genoma del ratto ha utilizzato ZFNs appositamente progettate per causare rotture nel ds-DNA che contengono mutazioni dal meccanismo di riparazione del DNA impreciso (da NHEJ) e quindi conteneva mutazioni specifiche (Geurts, et al. 2009). Questo processo, �nockout del gene,� impedisce la produzione della proteina normalmente trascritta dal gene bersaglio. Sono state utilizzate ZFN a cinque e sei dita per ottenere un alto livello di specificità nel legame mirato al gene per tre diverse proteine: proteina fluorescente verde (GFP), immunoglobulina M (IgM) e Rab38. Il knockout ha avuto successo nel 12% dei ratti testati; questi animali non avevano alcuna proteina wild-type e nessuna espressione. Le ZFN erano sufficientemente specifiche che non sono state osservate mutazioni in nessuno dei 20 siti non bersaglio previsti. Questo studio supporta la fattibilità del controllo della trascrizione e dell’espressione per il trattamento della malattia e l’importanza del legame specifico.
Abbiamo visto che due fattori principali contribuiscono al riconoscimento specifico del DNA da parte delle proteine; la formazione di legami idrogeno a specifici donatori e accettori di nucleotidi nel solco maggiore, e deformazioni dipendenti dalla sequenza dell’elica del DNA in forme alterate con maggiore affinità dei ligandi proteici. Per esempio la Tata Binding Protein (TBP) può interagire con un solco minore allargato nella scatola TATA. Nuove scoperte sostengono che inoltre le proteine sono in grado di utilizzare le informazioni nei solchi minori che sono diventati “ristretti” a seconda della sequenza nucleotidica.
Le tracce di DNA arricchite in A possono portare a conformazioni tortuose che causano legami idrogeno inter-base-pair nei solchi maggiori, risultati nel restringimento dei solchi minori. Elevate quantità di coppie di basi AT sono concentrate nei solchi minori stretti (larghezza <5,0 �) e le coppie di basi CG si trovano più frequentemente nei solchi minori larghi.
In che modo il restringimento dei solchi minori influenza il riconoscimento del DNA? I solchi minori stretti aumentano il potenziale elettrostatico negativo del DNA, rendendolo un sito più specifico e riconoscibile. I fosfati della spina dorsale del DNA sono più vicini al centro del solco quando è stretto, correlando così i solchi minori stretti con un potenziale elettrostatico più negativo.
Le parti che interagiscono con il solco minore delle proteine contengono arginina la cui catena laterale può essere alloggiata nel solco minore più stretto e negativo. Le arginine possono legarsi e in alcuni casi inserirsi come brevi motivi di sequenza che aumentano la specificità del riconoscimento della forma del DNA. L’Arg è preferito al Lys poiché il raggio effettivo della carica nell’Arg è maggiore di quello del portatore di carica nel Lys. Questo porterebbe ad una diminuzione dell’energia di desolvatazione per l’Arg che promuoverebbe il suo legame al solco maggiore ristretto. Questa scoperta dimostra che “il ruolo della forma del DNA deve essere preso in considerazione quando si annota l’intero genoma e si predicono i siti di legame dei fattori di trascrizione”.
Figura: Arg nel legame della trasposasi T3c nel boschetto minore ristretto del trasposone T3c
- Modello scorrevole per le interazioni proteine/DNA
Contribuenti e attribuzioni
- Prof. Henry Jakubowski (College of St. Benedict/St. John’s University)