Polimorfismo a singolo nucleotide
Il polimorfismo a singolo nucleotide (SNP) si riferisce alla differenza del singolo nucleotide nella stessa posizione nella sequenza genomica del DNA. In generale, una variazione di un singolo nucleotide con una frequenza superiore all’1% è chiamata SNP. Gli SNP comportano solo una variazione di una singola base, che può essere causata da una transizione o una trasversione di una singola base. C’è circa un SNP ogni 1000 basi nel genoma umano, e il numero totale di SNP nel genoma umano è circa 3 x 106.
SNP è diventato la terza generazione di marcatori genetici. Le caratteristiche degli SNP li rendono più adatti agli studi sulla pleiotropia genetica nei tratti complessi e nelle malattie, e al riconoscimento genico basato sulla popolazione rispetto ad altri marcatori polimorfici. Gli SNP hanno un’importanza significativa nella ricerca biomedica. Gli SNP sono stati utilizzati negli studi di associazione genome-wide come marcatori ad alta risoluzione nella mappatura dei geni relativi a malattie o tratti normali. Anche gli SNPs senza un impatto osservabile sul fenotipo (chiamati mutazioni silenziose) possono essere utili grazie alla loro quantità e alla loro eredità stabile nel corso delle generazioni.
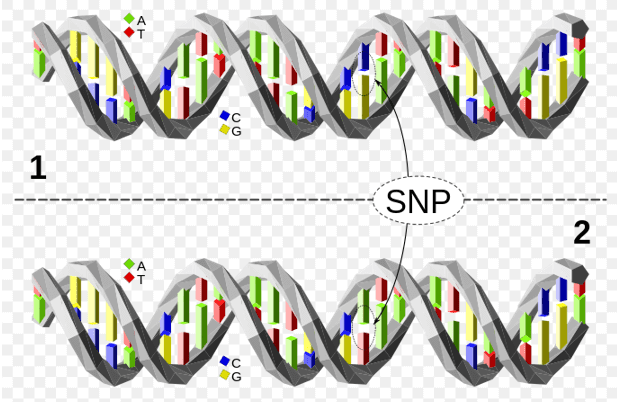 Figura 1. La molecola di DNA superiore differisce dalla molecola di DNA inferiore in una singola posizione di coppia di basi (un polimorfismo C/A).
Figura 1. La molecola di DNA superiore differisce dalla molecola di DNA inferiore in una singola posizione di coppia di basi (un polimorfismo C/A).
Microarray
Il microarray di DNA si riferisce a un chip genetico con un gran numero di sequenze di DNA sonda in una specifica disposizione immobilizzata su un substrato solido. Il principio è la teoria dell’ibridazione degli acidi nucleici, e il DNA campione rilevato viene ibridato con il microarray di DNA ed esteso. Successivamente, i frammenti della reazione di legame non complementare sul chip vengono lavati via, e il chip genico viene sottoposto a scansione laser confocale. Le intensità del segnale di fluorescenza sono poi misurate e interpretate come l’abbondanza di diversi geni attraverso una certa elaborazione dei dati.
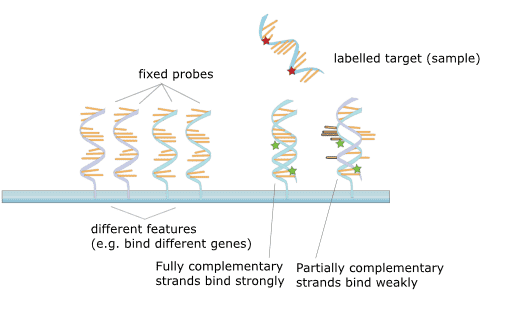 Figura 2. Il principio del microarray.
Figura 2. Il principio del microarray.
Microarray SNP
Il microarray SNP utilizza sequenze nucleotidiche note come sonde per ibridarsi con le sequenze di DNA esaminate, permettendo l’analisi qualitativa e quantitativa degli SNP attraverso la rilevazione del segnale. Rispetto al tradizionale metodo diagnostico a cella singola, il microarray SNP è un metodo ad alta produttività, che è in grado di eseguire migliaia di reazioni sulla superficie del chip oligonucleotidico in una sola volta.
Il flusso di lavoro del microarray SNP
Il flusso di lavoro generale del microarray SNP è mostrato nella figura 3. In breve, ci sono diversi processi: Fabbricazione del chip SNP, preparazione del DNA genomico del campione, ibridazione e scansione a fluorescenza.
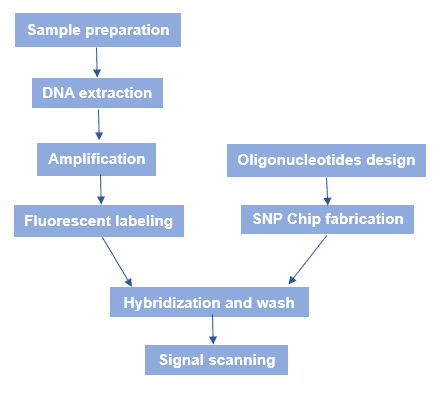 Figura 3. Il flusso di lavoro del microarray SNP.
Figura 3. Il flusso di lavoro del microarray SNP.
- Fabbricazione di chip SNP
I chip di oligonucleotidi o cDNA predefiniti sono disposti in modo ordinato e ad alta densità su un supporto di vetro per creare microarray o core chip. I metodi di fabbricazione del chip includono la sintesi in situ guidata dalla luce, il metodo dello spray chimico, il metodo del rivestimento di punti di contatto, la sintesi controllata del DNA in situ, il metodo di stampa micromeccanica senza contatto TOPSPOT® e la riproduzione con litografia morbida, ecc. Un totale di 4×105 diverse molecole di DNA può ora essere collocato su un chip di 1 cm2. La tecnologia Affymetrix GeneChip® permette la sintesi di oligonucleotidi in situ ad alta densità, ed è stata un pioniere in questo campo. - Preparazione del DNA genomico del campione
Prima di tutto, la preparazione del materiale campione. Poi il DNA genomico viene estratto e viene amplificato tramite PCR. Infine, vengono utilizzati diversi coloranti fluorescenti per etichettarlo. - Ibridizzazione e scansione
Il DNA genomico etichettato con fluorescenza viene ibridato con il microarray SNP in condizioni di reazione adeguate. Dopo essere stato lavato, uno scanner di fluorescenza specifico viene utilizzato per scansionare la fluorescenza. Poi l’analisi dell’immagine al computer viene fatta per elaborare i dati prima che venga effettuata l’analisi bioinformatica del gene bersaglio.
A CD Genomics, ci dedichiamo a fornire servizi di genotipizzazione SNP affidabili, compresa la genotipizzazione tramite sequenziamento, microarray SNP, genotipizzazione SNP MassARRAY, Hi-SNPseq, SNaPshot Multiplex System per la genotipizzazione SNP, e genotipizzazione TaqMan SNP.
- Iwamoto K, Bundo M, Ueda J, et al. Rilevamento di alterazioni strutturali cromosomiche in singole cellule da array SNP: un’indagine sistematica di bias di amplificazione e flusso di lavoro ottimizzato. Plos One, 2007, 2(12): e1306.
- Ho C C, Mun K S, Naidu R. SNP array technology: an array of hope in breast cancer research. Malaysian Journal of Pathology, 2013, 35(1):33-43.
- Ahmad A, Iqbal M A. Significato dell’analisi genome-wide delle alterazioni del numero di copie e UPD nelle sindromi mielodisplastiche utilizzando combinato CGH-SNP array. Current Medicinal Chemistry, 2012, 19(22).
- Sund K L, Zimmerman S L, Thomas C, et al. Regioni di omozigosi identificati da SNP microarray analisi aiuto nella diagnosi di malattia autosomica recessiva e incidentalmente rilevare rapporti di sangue dei genitori. Genetics in Medicine Official Journal of the American College of Medical Genetics, 2013, 15(1):70-78.
- Liu S, Zhou Z, Lu J, et al. Generazione di SNPs associati al gene su scala genomica nel pesce gatto per la costruzione di un array SNP ad alta densità. Bmc Genomics, 2011, 12(1):53.