一塩基多型
一塩基多型(SNP)とは、ゲノムDNA配列中の同じ位置の一塩基が異なることを指します。 一般に、1%以上の頻度で存在する一塩基の変異をSNPと呼ぶ。 SNPは一塩基の変異のみを含み、一塩基の転移やトランスバージョンによって引き起こされることがある。 ヒトゲノムには1000塩基あたり約1個のSNPが存在し、その総数は約3×106個である。
SNPは第3世代の遺伝マーカーとなった。 SNPの特徴は、他の多型マーカーに比べて、複雑な形質や疾患における遺伝的多面性の研究、集団ベースの遺伝子認識などに適していることである。 SNPsは生物医学研究において大きな重要性を持っている。 SNPはゲノムワイド関連研究において、疾患や正常形質に関連する遺伝子マッピングの高解像度マーカーとして利用されてきた。 また、表現型への影響を観察できないSNP(サイレントミューテーションと呼ばれる)は、その量や世代を超えて安定的に遺伝することから、有用であると考えられる。
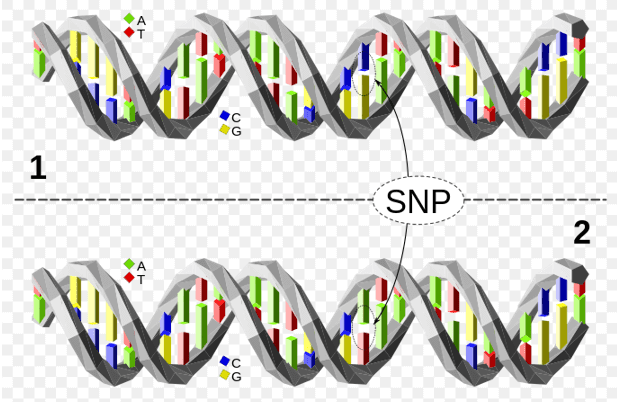 Fig. 上のDNA分子は下のDNA分子と1塩基対の位置が異なる(C/A多型)
Fig. 上のDNA分子は下のDNA分子と1塩基対の位置が異なる(C/A多型)
マイクロアレイ
DNAマイクロアレイとは、固体基板上に特定の配列で多数のプローブDNA配列を固定化した遺伝子チップのことである。 原理は核酸ハイブリダイゼーション理論であり、検出された試料DNAはDNAマイクロアレイとハイブリダイズして伸長する。 その後、チップ上の非相補的結合反応の断片を洗浄し、遺伝子チップをレーザーコンフォーカルスキャンする。 その後、蛍光シグナル強度を測定し、一定のデータ処理により異なる遺伝子の存在量として解釈する。
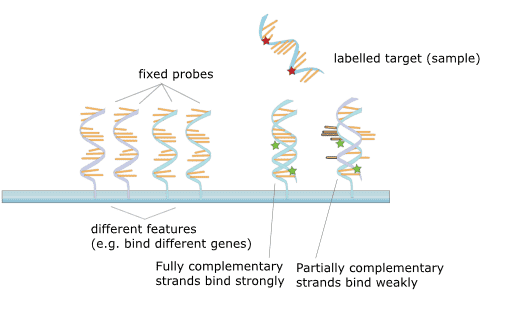 図2. マイクロアレイの原理
図2. マイクロアレイの原理
SNPマイクロアレイ
SNPマイクロアレイは、既知の塩基配列をプローブとして用い、被検DNA配列とハイブリダイズし、シグナル検出による定性・定量SNP解析が可能である。 従来のシングルセル診断法と比較して、SNPマイクロアレイはオリゴヌクレオチドチップの表面で一度に数千の反応を行うことができるハイスループット法である。
SNPマイクロアレイのワークフロー
SNPマイクロアレイの一般的なワークフローを図3に示した。 簡単に言えば、いくつかの工程がある。 SNPチップの作製、サンプルゲノムDNAの調製、ハイブリダイゼーション、蛍光スキャンである。
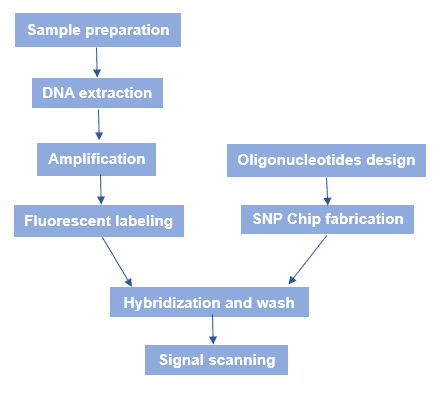 Figure 3. SNPマイクロアレイのワークフロー
Figure 3. SNPマイクロアレイのワークフロー
- SNPチップの作製
あらかじめデザインしたオリゴヌクレオチドやcDNAチップをガラス担体に整然と高密度に配列してマイクロアレイやコアチップを作製している。 チップの作製法としては、光誘導in-situ合成法、化学スプレー法、接触ドットコーティング法、in-situ DNA制御合成法、非接触マイクロメカニカル印刷法TOPSPOT®、ソフトリソグラフィー再生法など。 1cm2のチップに合計4×105種類のDNA分子を配置することが可能になりました。 Affymetrix社のGeneChip®テクノロジーは、高密度でin situのオリゴヌクレオチド合成を可能にし、この分野のパイオニアとなっています。 - サンプルゲノムDNA調製
まず、サンプル物質を調製します。 次に、ゲノムDNAを抽出し、PCRで増幅する。 3307> - ハイブリダイゼーションとスキャン 蛍光標識したゲノムDNAを適切な反応条件下でSNPマイクロアレイにハイブリダイズさせます。 洗浄後、特定の蛍光スキャナーで蛍光をスキャンする。
CDジェノミクスでは、シーケンサーによるジェノタイピング、SNPマイクロアレイ、MassARRAY SNP genotyping、Hi-SNPseq、SNaPshot Multiplex System for SNP genotyping, TaqMan SNP genotypingなどの信頼できるSNP遺伝子型判定サービスの提供に取り組んでいます。
- Iwamoto K, Bundo M, Ueda J, et al. Detection of chromosomal structural alterations in single cells by SNP arrays: a systematic survey of amplification bias and optimized workflow.SNPアレイによる単一細胞の染色体構造変化の検出:増幅バイアスと最適化されたワークフローに関するシステマティックな調査。 Plos One, 2007, 2(12): e1306.
- Ho C C, Mun K S, Naidu R. SNP array technology: an array of hope in breast cancer research.「SNPアレイ技術:乳がん研究における希望のアレイ」.
- Ahmad A, Iqbal M A. Significance of genome-wide analysis of copy number alterations and UPD in myelodysplastic syndromes using combined CGH-SNP arrays.マレーシア病理学雑誌, 2013, 35(1):33-43.
- Ahmad A, Iqbal M A. SNPアレイの意義は? SNPマイクロアレイ解析により同定されたホモ接合領域は常染色体劣性疾患の診断に役立ち、また親の血縁関係を偶発的に検出する。 Genetics in Medicine Official Journal of the American College of Medical Genetics, 2013, 15(1):70-78.
- Liu S, Zhou Z, Lu J, et al. Generation of genome-scale gene-associated SNPs in catfish for construction of a high-density SNP array.ナマズにおけるゲノム規模の遺伝子関連SNPの生成。 Bmc Genomics, 2011, 12(1):53.
。