Editorial on the Research Topic
The Role of AAA+ Proteins in Protein Repair and Degradation
ATPases Associated with diverse cellular Activities (AAA+) omvatten een superfamilie van eiwitten die een grote verscheidenheid van functies vervullen die essentieel zijn voor de celfysiologie, waaronder controle van eiwithomeostase, DNA replicatie, recombinatie, chromatine remodellering, ribosomaal RNA verwerking, moleculaire targeting, organel biogenese, en membraanfusie (Hanson en Whiteheart, 2005; Erzberger en Berger, 2006; Snider et al., 2008). Leden van deze superfamilie worden gedefinieerd door de aanwezigheid van het zogenaamde AAA+ domein dat de canonieke Walker A- en B-motieven bevat die nodig zijn voor ATP-binding en -hydrolyse (Hanson en Whiteheart, 2005). Doorgaans coderen genomen op ongeveer tien tot enkele honderden AAA+ familieleden (tabel 1; Finn et al., 2017), waarvan elk geacht wordt te zijn aangepast aan specifieke functionele niches die precieze mechanismen van substraatherkenning en -verwerking vereisen (Hanson en Whiteheart, 2005). De opvallende adaptieve radiatie van AAA+ eiwitten om in diverse settings te opereren, illustreert het veelzijdige nut van het AAA+ domein (Erzberger en Berger, 2006). AAA+ eiwitten vormen meestal hexamere complexen en fungeren als motoren om andere eiwitten, DNA/RNA, of multicomponent complexen te remodelleren (Figuur 1). Inderdaad, veel chaperones en ATP-afhankelijke proteasen zijn of hebben subeenheden die tot deze superfamilie behoren (figuur 1; Olivares et al., 2016).
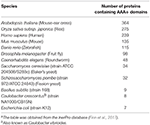
Tabel 1. Aantal AAA+ eiwitten in modelorganismena.
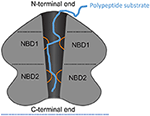
Figuur 1. Schematische weergave van ClpA als voorbeeld van een AAA+ hexamer. Schematische weergave van de domeinarchitectuur en interacties van ClpA als voorbeeld AAA+ hexamer met twee AAA+ domeinen per monomeer. Hier wordt een zijaanzicht van de ClpA hexamer getoond om het centrale, polypeptide-geleidende kanaal af te beelden. ClpA bevat drie domeinen, waaronder een N-terminaal domein en twee AAA+ domeinen: nucleotide-bindend domein 1 en 2 (NBD1 en NBD2). Het N-terminale domein interageert met regulatoren van de substraatspecificiteit, terwijl het C-terminale uiteinde interageert met het gekamerde protease ClpP. Mobiele lussen van NBD1 en NBD2 (oranje) projecteren in het centrale kanaal en grijpen polypeptide substraat (blauw) waardoor koppeling van ATP hydrolyse aan polypeptide translocatie over het centrale kanaal mogelijk wordt.
De afgelopen jaren is er aanzienlijke vooruitgang geboekt bij het identificeren van de structuur en het functionele mechanisme van een groot aantal AAA+ eiwitten (Gates et al., 2017; Puchades et al., 2017; Ripstein et al., 2017; Zehr et al., 2017). In dit onderzoeksthema worden verschillende elementen van deze opwindende vooruitgang overgebracht in 21 artikelen, die een gedetailleerd structureel en mechanistisch beeld omvatten van verschillende AAA+ chaperones en proteases, waaronder: ClpX (Alhuwaider en Dougan; Bittner et al.; Elsholz et al.; LaBreck et al.; Vass et al.), ClpA (Bittner et al.; Duran et al.), ClpB en Hsp104 (Chang et al.Duran et al.; Franke et al.; Johnston et al.), Hsp78 (Abrahão et al.), ClpC (Alhuwaider en Dougan; Elsholz et al.), ClpE (Elsholz et al.), Pontin (Mao en Houry), Reptin (Mao en Houry), FtsH (Alhuwaider en Dougan), 19S proteasoom (Snoberger et al.; Yedidi et al.), Lon (Alhuwaider en Dougan; Bittner et al.; Fishovitz et al.), p97 (Hänzelmann en Schindelin; Saffert et al.; Ye et al.), Pex1/6 (Saffert et al.), CbbQ (Mueller-Cajar), rubisco activase (Bhat et al.), torsines (Chase et al.), en mitochondriale AAA+ proteasen (Glynn). Hier introduceren we deze fascinerende werken.
Studies over ClpXP, Lon, en verwante ATP-afhankelijke Proteasen
In hun onderzoeksartikel, “The Protein Chaperone ClpX Targets Native and Non-native Aggregated Substrates for Remodeling, Disassembly, and Degradation with ClpP,” voeren LaBreck et al. een reeks elegante experimenten uit om vast te stellen dat ClpX disaggregase-activiteit bezit tegen polypeptiden die specifieke ClpX-herkenningssignalen bevatten (LaBreck et al.). In aanwezigheid van ClpP koppelt ClpX disaggregatie van deze substraten aan hun degradatie. Belangrijk is dat zij ook vaststellen dat ClpXP de accumulatie voorkomt van aggregaten gevormd door eiwitten met ClpX-herkenningssignalen in vivo (LaBreck et al.). Deze studies belichten ClpX als een eiwitsplitser, die voorheen ondergewaardeerd was.
In hun onderzoeksartikel, “The Essential Role of ClpXP in Caulobacter crescentus Requires Species Constrained Substrate Specificity,” onderzoeken Vass et al. soortspecifieke functies van ClpX (Vass et al.). Vreemd genoeg is ClpX essentieel in sommige soorten zoals C. crescentus, maar niet essentieel in andere bacteriën zoals E. coli (Vass et al.). Belangrijk is dat E. coli ClpX niet in staat was C. crescentus ClpX in vivo aan te vullen (Vass et al.). Dit gebrek aan activiteit was te wijten aan soortspecifieke verschillen in het N-terminale domein van ClpX, die van cruciaal belang zijn voor de verwerking van de replicatieklem-lader-subeenheid DnaX in C. crescentus. Kleine verschillen in ClpX specificiteit kunnen dus bijzonder kritisch zijn voor specifieke bacteriesoorten.
In hun review over “Functional Diversity of AAA+ Protease Complexes in Bacillus subtilis,” bespreken Elsholz et al. de functies van verschillende AAA+ proteasen in B. subtilis, te weten: ClpCP, ClpEP, ClpXP, ClpYQ, LonA/B, en FtsH (Elsholz et al.). Zij bespreken hoe verschillende stressreacties hun expressie controleren en de fenotypes die worden waargenomen bij verwijdering van deze verschillende proteasen. Het vermogen van sommige van deze proteasen om competentie, sporulatie, motiliteit en biofilmvorming te controleren wordt beschreven. Tenslotte bespreken de auteurs het richten van deze proteasen voor de ontwikkeling van nieuwe antibiotica.
In hun review getiteld “AAA+ Machines of Protein Destruction in Mycobacteria,” bespreken Alhuwaider et al. (Alhuwaider en Dougan) recente vorderingen bij het bepalen van de structuur en functie van AAA+ proteasen van mycobacteria. Deze proteasen zijn: ClpXP1P2, ClpC1P1P2, Lon, FtsH, en Mpa. De auteurs bespreken ook het Pup-proteasoom systeem (PPS) dat aanwezig is in mycobacteriën en dat equivalent is aan het ubiquitine-proteasoom systeem in eukaryoten. Alhuwaider et al. besluiten met een bespreking van nieuwe verbindingen die de activiteit van ClpP1P2 ontregelen of remmen en andere die ClpC1 ontregelen. Deze verbindingen hebben veelbelovende activiteiten tegen mycobacteriën.
In een onderzoeksartikel getiteld “The Copper Efflux Regulator CueR Is Subject to ATP-Dependent Proteolysis in Escherichia coli,” tonen Bittner et al. aan dat de AAA+ proteasen Lon, ClpXP, en ClpAP verantwoordelijk zijn voor de degradatie van E. coli CueR, dat een transcriptiefactor is die de inductie van het koper efflux Cue systeem regelt (Bittner et al.). De auteurs stelden vast dat de herkenning van CueR door de AAA+ proteasen de toegankelijke C-terminus van CueR vereist. Zij concluderen dat ATP-afhankelijke proteasen nodig zijn voor koperhomeostase in E. coli.
Fishovitz et al. voeren een gedetailleerde vergelijking uit tussen menselijk en E. coli Lon in hun onderzoeksartikel getiteld “Utilization of Mechanistic Enzymology to Evaluate the Significance of ADP Binding to Human Lon Protease” (Fishovitz et al.). Met behulp van een gedetailleerde mechanistische studie ontdekten zij dat menselijk Lon, in tegenstelling tot E. coli Lon, een lage affiniteit heeft voor ADP, ondanks het feit dat het vergelijkbare kcat- en KM-waarden vertoont in de ATPase-activiteit. Zij stellen voor dat het menselijke Lon niet wordt gereguleerd door een substraat-gepromoteerd ADP/ATP uitwisselingsmechanisme. Deze verschillen tussen menselijk en E. coli Lon zouden de toekomstige ontwikkeling van soortspecifieke Lon-remmers mogelijk kunnen maken.
In zijn review over “Multifunctionele Mitochondriale AAA Proteasen,” bespreekt Dr. Glynn de twee mitochondriale AAA proteasen, i-AAA en m-AAA (Glynn). Beide zijn mitochondriale binnenmembraaneiwitten. Echter, i-AAA projecteert de ATPase en protease domeinen in de mitochondriale intermembraan ruimte, terwijl het m-AAA protease de katalytische domeinen in de matrix projecteert. De structuren van deze proteasen worden besproken, evenals hun werkingsmechanisme. De proteasen kunnen volledige substraatafbraak uitvoeren, maar kunnen ook alleen bepaalde substraten klieven, zoals voor MrpL32 en Atg32.
ClpB en Hsp104
In hun mini-review, “Structural Elements Regulating AAA+ Protein Quality Control Machines,” Chang et al. besproken hoe de pore-lus-1, het Inter-Subunit Signaling motief, en het Pre-Sensor I insert motief zouden kunnen bijdragen aan de activiteit van twee Hsp100 disaggregases, bacteriële ClpB en gist Hsp104 (Chang et al.). Zij stellen een model voor hoe deze structurele elementen de AAA+ ATPase cyclus zouden kunnen koppelen aan substraat translocatie door het centrale kanaal van ClpB en Hsp104. Dit proces van polypeptide translocatie wordt verondersteld ten grondslag te liggen aan hoe ClpB en Hsp104 polypeptiden uit geaggregeerde structuren halen (Chang et al.).
Duran et al. geven een “Vergelijkende Analyse van de Structuur en Functie van AAA+ Motoren ClpA, ClpB, en Hsp104: Gemeenschappelijke Draden en Ongelijksoortige Functies” in hun review (Duran et al.). Zij bespreken het vermogen van deze drie AAA+ eiwitten (ClpA, ClpB, en Hsp104) om polypeptiden te transloceren door hun hexamere complexen. Al deze eiwitten hebben twee AAA+ domeinen en zijn bekend voor het ontvouwen van eiwitten. Belangrijk is dat van ClpB en Hsp104 ook bekend is dat zij fungeren als disaggregases, terwijl ClpA een complex kan vormen met het ClpP protease. De auteurs wijzen op de noodzaak van het gebruik van transient state kinetische methoden om de kinetische mechanismen van deze motoreiwitten te onderzoeken. Zij beschrijven hoe het gebruik van dergelijke methoden hen in staat stelde aan te tonen dat, bijvoorbeeld, ClpA polypeptiden transporteert met ongeveer 20 aa s-1, terwijl in complex met het ClpP protease, de ClpA translocatiesnelheid nog hoger is met ongeveer 35 aa s-1. De auteurs bespreken ook het belang van het chaperon Hsp70 in de functie van ClpB/Hsp104, en de waarneming van soortspecificiteit in de interactie tussen Hsp70 en ClpB/Hsp104.
In hun onderzoeksartikel getiteld “Mutant Analysis Reveals Allosteric Regulation of ClpB Disaggregase,” voeren Franke et al. mutatie-analyses uit op het E. coli ClpB disaggregase om de allosterische regulatie ervan te karakteriseren (Franke et al.). ClpB kan worden onderverdeeld in een N-terminaal domein en twee AAA+ domeinen gescheiden door een spiraalvormig gebied dat het M-domein wordt genoemd. De auteurs identificeren een zeer geconserveerd residu in het eerste AAA+ domein, A328. De ClpB-A328V mutant bleek een zeer hoge ATPase activiteit te hebben en vertoonde cellulaire toxiciteit. Onverwacht was dat de hoge ATPase activiteit van ClpB-A328V voornamelijk te wijten was aan de tweede AAA+ ring zoals beoordeeld met amide waterstof uitwisseling massaspectrometrie. De auteurs concluderen dat A328 een cruciaal residu is in de controle van de ATP hydrolyse in beide AAA+ ringen van ClpB.
In hun onderzoeksartikel getiteld “Substrate Discrimination by ClpB and Hsp104,” beschrijven Johnston et al. de aangeboren substraatvoorkeuren van ClpB en Hsp104 in de afwezigheid van de DnaK en Hsp70 chaperon systemen (Johnston et al.). Zij tonen aan dat de substraatspecificiteit wordt bepaald door het eerste AAA+ domein in elk eiwit. Zij kwamen tot deze conclusie door de twee chaperones te testen op hun vermogen om op verschillende modelsubstraten in te werken. Zij testten ook verschillende chimaera’s van de twee chaperones.
In “Hsp78 (78 kDa Heat Shock Protein), a Representative AAA Family Member Found in the Mitochondrial Matrix of Saccharomyces cerevisiae,” bespreken Abrahão et al. de structuur en functie van Hsp78 (Abrahao et al.). Hsp78 is de mitochondriale paraloog van Hsp104, dat functioneert bij eiwitontbinding en reactivering (Abrahao et al.). Vreemd genoeg zijn Hsp104 en Hsp78 verloren gegaan bij de overgang van protozoa naar metazoa (Abrahao et al.). Abrahao et al. bespreken echter het bestaan van ANKCLP, dat naast Hsp78 en Hsp104 in protozoa verschijnt en de evolutionaire overgang naar metazoa overleeft. ANKCLP bezit een AAA+ domein dat lijkt op nucleotide-bindend domein 2 (NBD2) van Hsp104 en Hsp78, maar is voor het overige sterk verschillend. Intrigerend genoeg veroorzaken mutaties in ANKCLP 3-methylglutaconzuururie, progressieve hersenatrofie, verstandelijke handicap, congenitale neutropenie, cataract en bewegingsstoornissen bij de mens (Abrahao et al.).
p97
In hun review, “Structure and Function of p97 and Pex1/6 Type II AAA+ Complexes,” bespreken Saffert et al. twee verschillende AAA+ complexen die ubiquitinated substrate proteins remodelleren (Saffert et al.). Eén functie van p97 is het disloceren van geübiquitineerde substraten van het ER membraan naar het proteasoom tijdens ER-geassocieerde degradatie (Saffert et al.). Pex1/Pex6 daarentegen is een heterohexamerische motor die bestaat uit afwisselend Pex1 en Pex6 subeenheden, en die essentieel is voor peroxisome biogenese en functie. Recente cryo-elektronenmicroscopie (cryo-EM) structuren van p97 en Pex1/6 worden besproken en belangrijke structurele verschillen worden belicht.
In hun review getiteld “A Mighty ‘Protein Extractor’ of the Cell: Structure and Function of the p97/CDC48 ATPase,” vatten Ye et al. de huidige kennis van de structuur en functie van p97 en zijn rol in verschillende ziekten samen (Ye et al.). p97 heeft twee AAA+ domeinen die met een korte linker zijn verbonden. Het heeft ook een N-terminaal domein, dat zijn interacties met verschillende adaptoreiwitten medieert. De auteurs geven een gedetailleerde bespreking van de structuur van p97 en het effect van nucleotiden op zijn verschillende conformatie. Deze studies zijn gebaseerd op het gebruik van technieken zoals EM, X-straal kristallografie, en hoge-snelheid atomaire kracht microscopie. De auteurs bespreken vervolgens de multicellulaire functies van dit sterk geconserveerde eiwit, waaronder zijn rol in ER-geassocieerde eiwitafbraak (ERAD), mitochondrie-geassocieerde afbraak (MAD) door polypeptiden te extraheren uit het mitochondriale buitenmembraan, en ribosoom-geassocieerde afbraak (RAD). Tenslotte geven Ye et al. een overzicht van p97-mutaties die leiden tot verschillende menselijke ziekten zoals IBMPFD (Inclusion Body Myopathy associated with Paget’s disease of the bone and Frontotemporal Dementia), FALS (familiaire amyotrofische laterale sclerose), CMT2Y (Charcot-Marie-Tooth disease, type 2Y), hereditaire spastische paraplegieën (HSP), de ziekte van Parkinson (PD), en de ziekte van Alzheimer (AD).
In hun review over p97 getiteld “The Interplay of Cofactor Interactions and Post-translational Modifications in the Regulation of the AAA+ ATPase p97,” bespreken Hänzelmann en Schindelin hoe verschillende cofactoren de activiteit van het p97 ATPase moduleren (Hanzelmann en Schindelin). Zij benadrukken het feit dat het vermogen van p97 om betrokken te zijn bij een groot aantal cellulaire processen te danken is aan het grote aantal cofactoren dat met dit eiwit interageert. Zij hebben drie verschillende klassen van p97 cofactoren opgehelderd, namelijk: (i) Substraat-recruiting cofactoren zoals UBA-UBX eiwitten en UFD1-NPL4, (ii) Substraat-verwerkende cofactoren zoals ubiquitine (E3) ligases en deubiquitinases (DUBs), en (iii) Regulerende cofactoren zoals de UBX eiwitten, die p97 hexamers kunnen sequestreren of recyclen. De auteurs bespreken ook de rol van post-translationele modificaties op p97 activiteit, en op zijn interacties met zijn cofactoren en substraten.
AAA+ Proteïnen van het Proteasoom
In “AAA-ATPases in Protein Degradation,” Yedidi et al. de activiteiten van Rpt1, Rpt2, Rpt3, Rpt4, Rpt5, en Rpt6, die de AAA+ ATPasen van het eukaryotische proteasoom zijn, evenals enkele van hun bacteriële verwanten zoals PAN, Mpa, en VAT (Yedidi et al.). Zij richten zich op nieuwe technologieën om te begrijpen hoe deze AAA+ ATPasen functioneren door ongevouwen polypeptiden te transloceren naar de proteolytische kamer van het protease (Yedidi et al.). Conformationele veranderingen binnen de AAA+ ring en de aangrenzende protease-kamer lijken een peristaltisch pompmechanisme op te wekken om substraten af te leveren voor afbraak (Yedidi et al.).
In hun onderzoeksartikel, “The Proteasomal ATPases Use a Slow but Highly Processive Strategy to Unfold Proteins,” stellen Snoberger et al. vast dat proteasomale AAA+ proteïnen een motormechanisme met lage snelheid maar zeer procesmatig gebruiken om substraten af te leveren aan de proteolytische holte van het proteasoom (Snoberger et al.). Dit mechanisme staat in contrast met ClpX, dat een motormechanisme gebruikt met een hoge snelheid, maar minder procesmatig, om substraten af te leveren aan het ClpP protease voor degradatie. Deze verschillen in motormechanismen kunnen geëvolueerd zijn in antwoord op verschillende eisen van hun specifieke clientèle.
Rubisco Activases
In hun review over “Rubisco Activases: AAA+ Chaperones Adapted to Enzyme Repair” bespreken Bhat et al. de unieke functie van het Rubisco activase (Rca) bij het remodelleren van Rubisco (Bhat et al.). Rca is een AAA+ chaperon dat in hoge mate geconserveerd is in fotosynthetische organismen, van bacteriën tot hogere planten. Rubisco is het enzym ribulose-1,5-bisfosfaatcarboxylase/oxygenase, dat betrokken is bij de vastlegging van CO2 uit de atmosfeer tijdens de fotosynthese. Het is het eiwit dat het meest voorkomt op aarde en is het sleutelenzym in de synthese van alle organische materie op de planeet. Rubisco is echter een arm enzym en wordt gemakkelijk geremd door nevenproducten van zijn katalytische reacties of door verbindingen die door sommige planten bij weinig licht worden gesynthetiseerd. Rca heeft tot taak Rubisco van dergelijke problematische remmingen te verlichten of te “genezen”. De auteurs bespreken de structuur van Rca van verschillende soorten en de mogelijke mechanismen van zijn functie.
Dr. Mueller-Cajar geeft een overzicht van “The Diverse AAA+ Machines that Repair Inhibited Rubisco Active Sites” (Mueller-Cajar). Hij bespreekt de aanwezigheid van drie evolutionair verschillende klassen van Rubisco-activases (Rcas): (1) groene en (2) rode Rcas die vooral voorkomen in fotosynthetische eukaryoten van respectievelijk de groene en rode plastid lineage, en (3) CbbQO die voorkomen in chemo-autotrofe bacteriën. Hij bespreekt de evolutie van deze activases en hun mogelijke gebruik in de synthetische biologie om de Rubisco-activiteit in planten te verbeteren.
Torsin
In hun perspectiefartikel, “Torsin ATPases: Harnessing Dynamic Instability for Function,” bespreken Chase et al. de Torsins, die ook fylogenetisch verwant zijn aan NBD2 van gist Hsp104 (Chase et al.). Torsins zijn de enige AAA+ ATPases die gelokaliseerd zijn binnen het ER en de verbonden nucleaire enveloppe (Chase et al.). Intrigerend is dat mutaties in TorsinA de oorzaak zijn van DYT1 dystonie, een neurologische aandoening bij de mens (Chase et al.). Torsines vertonen een zwakke ATPase activiteit die wordt verhoogd via active-site complementatie als gevolg van co-assemblage met specifieke accessoire cofactoren LAP1 en LULL1 (Chase et al.). Chase et al. suggereren dat dynamische assemblage en disassemblage van Torsin/cofactor complexen een belangrijke rol spelen in hun functie in nucleaire trafficking en nucleaire-porie complex assemblage (Chase et al.).
Pontin en Reptin
In hun uitgebreide review over “The Role of Pontin and Reptin in Cellular Physiology and Cancer Etiology,” bespreken Mao en Houry de vele functies van de zeer geconserveerde Pontin en Reptin AAA+ ATPases (Mao en Houry). Deze twee eiwitten functioneren gewoonlijk samen als een complex, maar kunnen ook onafhankelijk functioneren. De auteurs belichten de rol van Pontin en Reptin in de chromatine remodellering. Zij bespreken ook hoe Pontin en Reptin de transcriptionele activiteiten van verschillende proto-oncogenen zoals MYC en β-catenine moduleren. Mao en Houry verduidelijken hoe Pontin en Reptin nodig blijken te zijn voor de assemblage van PIKK signaal complexen en voor telomerase, mitotische spindel, RNA polymerase II, en snoRNPs. De auteurs besluiten met een overzicht van de huidige inspanningen gericht op het identificeren van remmers van Pontin en Reptin om te worden ontwikkeld als nieuwe antikankers.
Conclusie
In conclusie, deze verzameling van 21 artikelen belicht een aantal belangrijke structurele en mechanistische aspecten van AAA+ eiwitten betrokken bij eiwitherstel en -afbraak. We zijn enthousiast om te zien hoe het veld zich zal blijven ontwikkelen tijdens de aan de gang zijnde cryo-EM revolutie (Egelman, 2016). We verwachten dat cryo-EM een dieper begrip mogelijk zal maken van hoe deze fascinerende moleculaire machines werken in uiteenlopende situaties (Gates et al., 2017; Puchades et al., 2017; Ripstein et al., 2017; Zehr et al, 2017).
Author Contributions
Alle vermelde auteurs hebben een substantiële, directe en intellectuele bijdrage geleverd aan het werk, en hebben het goedgekeurd voor publicatie.
Conflict of Interest Statement
De auteurs verklaren dat het onderzoek is uitgevoerd in afwezigheid van enige commerciële of financiële relaties die zouden kunnen worden opgevat als een potentieel belangenconflict.
Acknowledgments
Het werk in het laboratorium van WH op dit gebied wordt gefinancierd door een Canadian Institutes of Health Research Project Grant (PJT-148564). JS wordt ondersteund door NIH grant R01GM099836.
Egelman, E. H. (2016). De huidige revolutie in Cryo-EM. Biophys. J. 110, 1008-1012. doi: 10.1016/j.bpj.2016.02.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Erzberger, J. P., and Berger, J. M. (2006). Evolutionaire verwantschappen en structurele mechanismen van AAA+ eiwitten. Annu. Rev. Biophys. Biomol. Struct. 35, 93-114. doi: 10.1146/annurev.biophys.35.040405.101933
PubMed Abstract | CrossRef Full Text | Google Scholar
Finn, R. D., Attwood, T. K., Babbitt, P. C., Bateman, A., Bork, P., Bridge, A. J., et al. (2017). InterPro in 2017 – verder dan eiwitfamilie- en domeinannotaties. Nucleic Acids Res. 45, D190-D199. doi: 10.1093/nar/gkw1107
PubMed Abstract | CrossRef Full Text | Google Scholar
Gates, S. N., Yokom, A. L., Lin, J., Jackrel, M. E., Rizo, A. N., Kendsersky, N. M., et al. (2017). Ratchet-like polypeptide translocation mechanism of the AAA+ disaggregase Hsp104. Science 357, 273-279. doi: 10.1126/science.aan1052
PubMed Abstract | CrossRef Full Text | Google Scholar
Hanson, P. I., and Whiteheart, S. W. (2005). AAA+ proteins: have engine, will work. Nat. Rev. Mol.Cell Biol. 6, 519-529. doi: 10.1038/nrm1684
PubMed Abstract | CrossRef Full Text | Google Scholar
Olivares, A. O., Baker, T. A., and Sauer, R. T. (2016). Mechanistische inzichten in bacteriële AAA+ proteasen en eiwit-remodelleringsmachines. Nat. Rev. Microbiol. 14, 33-44. doi: 10.1038/nrmicro.2015.4
PubMed Abstract | CrossRef Full Text | Google Scholar
Puchades, C., Rampello, A. J., Shin, M., Giuliano, C. J., Wiseman, R. L., Glynn, S. E., et al. (2017). Structuur van het mitochondriale binnenmembraan AAA+ protease YME1 geeft inzicht in substraatverwerking. Science 358:eaao0464. doi: 10.1126/science.aao0464
PubMed Abstract | CrossRef Full Text | Google Scholar
Ripstein, Z. A., Huang, R., Augustyniak, R., Kay, L. E., and Rubinstein, J. L. (2017). Structuur van een AAA+ unfoldase in het proces van ontvouwen van substraat. Elife 6:e25754. doi: 10.7554/eLife.25754
PubMed Abstract | CrossRef Full Text | Google Scholar
Snider, J., Thibault, G., and Houry, W. A. (2008). De AAA+ superfamilie van functioneel diverse eiwitten. Genome Biol. 9:216. doi: 10.1186/gb-2008-9-4-216
PubMed Abstract | CrossRef Full Text | Google Scholar
Zehr, E., Szyk, A., Piszczek, G., Szczesna, E., Zuo, X., and Roll-Mecak, A. (2017). Katanine spiraal- en ringstructuren werpen licht op vermogensslag voor microtubulusscheiding. Nat. Struct. Mol. Biol. 24, 717-725. doi: 10.1038/nsmb.3448
PubMed Abstract | CrossRef Full Text | Google Scholar