Single nucleotide polymorphism
Single nucleotide polymorphism (SNP) verwijst naar het verschil van één enkele nucleotide op dezelfde positie in de genomische DNA-sequentie. In het algemeen wordt een enkele nucleotide variatie met een frequentie van meer dan 1% een SNP genoemd. Bij SNP’s gaat het slechts om een enkele basevariatie, die kan worden veroorzaakt door een enkele baseovergang of transversie. Er is ongeveer één SNP per 1000 basen in het menselijk genoom, en het totale aantal SNP’s in het menselijk genoom bedraagt ongeveer 3 x 106.
SNP is de derde generatie van genetische markers geworden. De kenmerken van SNP’s maken ze geschikter voor studies naar genetische pleiotropie bij complexe eigenschappen en ziekten, en genherkenning op bevolkingsniveau dan andere polymorfe markers. SNP’s zijn van groot belang in het biomedisch onderzoek. SNP’s zijn in genoomwijde associatiestudies gebruikt als hoge-resolutie markers bij het in kaart brengen van genen in verband met ziekten of normale kenmerken. SNP’s zonder waarneembare invloed op het fenotype (stille mutaties genoemd) kunnen ook nuttig zijn vanwege hun hoeveelheid en stabiele overerving over generaties.
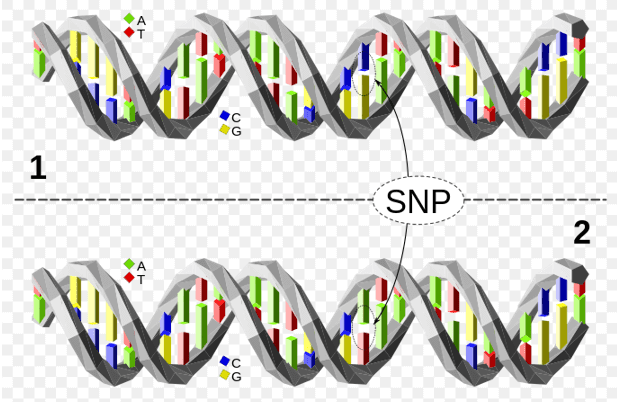 Figuur 1. Het bovenste DNA-molecuul verschilt van het onderste DNA-molecuul op een enkele basispaarlocatie (een C/A-polymorfisme).
Figuur 1. Het bovenste DNA-molecuul verschilt van het onderste DNA-molecuul op een enkele basispaarlocatie (een C/A-polymorfisme).
Microarray
De DNA-microarray verwijst naar een genchip met een groot aantal probe DNA-sequenties in een specifieke rangschikking, geïmmobiliseerd op een vast substraat. Het principe is de nucleïnezuur hybridisatie theorie, en het gedetecteerde monster DNA wordt gehybridiseerd met de DNA microarray en uitgebreid. Vervolgens worden de fragmenten van de niet-complementaire bindingsreactie op de chip weggespoeld, en wordt de genchip onderworpen aan laserconfocaal scannen. Fluorescentiesignaalintensiteiten worden vervolgens gemeten en geïnterpreteerd als de abundantie van verschillende genen via bepaalde gegevensverwerking.
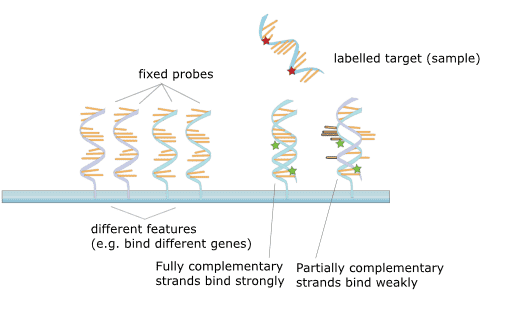 Figuur 2. Het principe van microarray.
Figuur 2. Het principe van microarray.
SNP microarray maakt gebruik van bekende nucleotide-sequenties als probes om te hybridiseren met de geteste DNA-sequenties, waardoor kwalitatieve en kwantitatieve SNP-analyse mogelijk wordt door signaaldetectie. Vergeleken met de traditionele diagnostische methode met één cel, is SNP-microarray een methode met hoge verwerkingscapaciteit, die in staat is om duizenden reacties op het oppervlak van de oligonucleotidechip tegelijk uit te voeren.
De workflow van SNP-microarray
De algemene workflow van SNP-microarray wordt weergegeven in figuur 3. Kort gezegd zijn er verschillende processen: SNP chip fabricage, monster genomisch DNA voorbereiding, hybridisatie, en fluorescentie scanning.
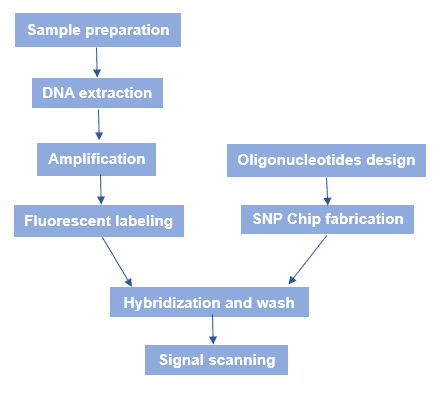 Figuur 3. De workflow van SNP-microarray.
Figuur 3. De workflow van SNP-microarray.
- Fabricage van SNP-chips
De vooraf ontworpen oligonucleotide- of cDNA-chips worden ordelijk en in hoge dichtheid op de glazen drager gerangschikt om microarrays of kernchips te maken. De fabricagemethoden van de chip omvatten lichtgeleide in-situ synthese, chemische spraymethode, contactpuntcoatingmethode, in-situ DNA gecontroleerde synthese, contactloze micromechanische drukmethode TOPSPOT® en zachte lithografische reproductie, enz. In totaal kunnen nu 4×105 verschillende DNA-moleculen op een chip van 1 cm2 worden geplaatst. De Affymetrix GeneChip®-technologie maakt oligonucleotidesynthese met hoge dichtheid in situ mogelijk en is op dit gebied een pionier geweest. - Monstervoorbereiding van genomisch DNA
Voreerst wordt het monstermateriaal voorbereid. Vervolgens wordt het genomisch DNA geëxtraheerd en door PCR geamplificeerd. Tenslotte worden verschillende fluorescerende kleurstoffen gebruikt om het te labelen. - Hybridisatie en scan
Het met fluorescentie gelabelde genomische DNA wordt onder geschikte reactieomstandigheden gehybridiseerd met SNP-microarray. Na te zijn gewassen, wordt een specifieke fluorescentiescanner gebruikt om de fluorescentie te scannen. Vervolgens wordt de computerbeeldanalyse gemaakt om de gegevens te verwerken voordat de bioinformatica-analyse van het doelgen wordt uitgevoerd.
Bij CD Genomics zijn we toegewijd aan het leveren van betrouwbare SNP-genotyperingsdiensten, waaronder genotypering door sequencing, SNP-microarray, MassARRAY SNP-genotypering, Hi-SNPseq, SNaPshot Multiplex System voor SNP-genotypering, en TaqMan SNP-genotypering.
- Iwamoto K, Bundo M, Ueda J, et al. Detection of chromosomal structural alterations in single cells by SNP arrays: a systematic survey of amplification bias and optimized workflow. Plos One, 2007, 2(12): e1306.
- Ho C C, Mun K S, Naidu R. SNP array technology: an array of hope in breast cancer research. Malaysian Journal of Pathology, 2013, 35(1):33-43.
- Ahmad A, Iqbal M A. Significance of genome-wide analysis of copy number alterations and UPD in myelodysplastic syndromes using combined CGH-SNP arrays. Current Medicinal Chemistry, 2012, 19(22).
- Sund K L, Zimmerman S L, Thomas C, et al. Regions of homozygosity identified by SNP microarray analysis aid in the diagnosis of autosomal recessive disease and incidentally detect parental blood relationships. Genetics in Medicine Official Journal of the American College of Medical Genetics, 2013, 15(1):70-78.
- Liu S, Zhou Z, Lu J, et al. Generation of genome-scale gene-associated SNPs in catfish for the construction of a high-density SNP array. Bmc Genomics, 2011, 12(1):53.