Polimorfizm pojedynczego nukleotydu
Polimorfizm pojedynczego nukleotydu (SNP) odnosi się do różnicy pojedynczego nukleotydu w tej samej pozycji w sekwencji genomowego DNA. Ogólnie rzecz biorąc, pojedyncza zmiana nukleotydu z częstotliwością większą niż 1% jest nazywana SNP. SNP obejmuje tylko pojedynczą zmianę zasad, która może być spowodowana przez pojedynczą zmianę zasad lub transwersję. W ludzkim genomie występuje w przybliżeniu jeden SNP na 1000 zasad, a całkowita liczba SNP w ludzkim genomie wynosi około 3 x 106.
SNP stały się markerami genetycznymi trzeciej generacji. Cechy SNP sprawiają, że są one bardziej odpowiednie do badań nad plejotropią genetyczną w złożonych cechach i chorobach oraz do populacyjnego rozpoznawania genów niż inne markery polimorficzne. SNP mają duże znaczenie w badaniach biomedycznych. SNP są wykorzystywane w badaniach asocjacyjnych obejmujących cały genom jako markery o wysokiej rozdzielczości w mapowaniu genów związanych z chorobami lub normalnymi cechami. SNP bez obserwowalnego wpływu na fenotyp (zwane cichymi mutacjami) mogą być również użyteczne ze względu na ich ilość i stabilne dziedziczenie przez pokolenia.
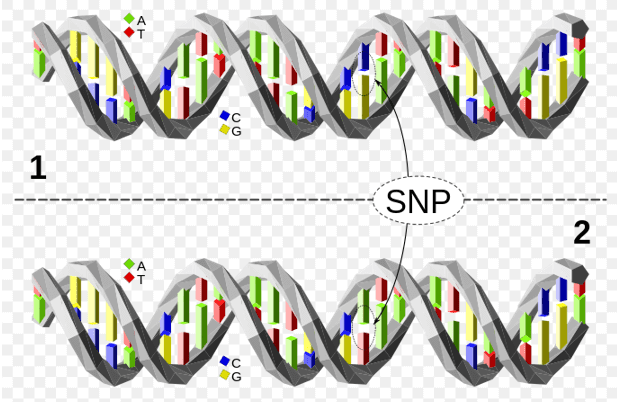 Ryc. 1. Górna cząsteczka DNA różni się od dolnej cząsteczki DNA w miejscu jednej pary zasad (polimorfizm C/A).
Ryc. 1. Górna cząsteczka DNA różni się od dolnej cząsteczki DNA w miejscu jednej pary zasad (polimorfizm C/A).
Mikromacierz
Mikromacierz DNA odnosi się do chipa genowego z dużą liczbą sekwencji DNA sondy w specyficznym układzie unieruchomionym na stałym podłożu. Zasadą jest teoria hybrydyzacji kwasów nukleinowych, a wykryte DNA próbki jest hybrydyzowane z mikromacierzą DNA i przedłużane. Następnie fragmenty niekomplementarnej reakcji wiązania na chipie są wypłukiwane, a chip genowy poddawany jest laserowemu skanowaniu konfokalnemu. Intensywności sygnału fluorescencji są następnie mierzone i interpretowane jako obfitość różnych genów poprzez pewne przetwarzanie danych.
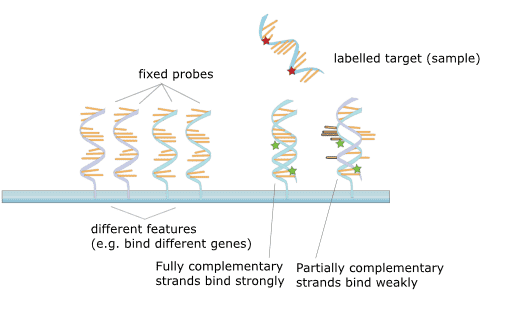 Figura 2. Zasada działania mikromacierzy.
Figura 2. Zasada działania mikromacierzy.
Mikromacierz SNP
Mikromacierz SNP wykorzystuje znane sekwencje nukleotydów jako sondy do hybrydyzacji z badanymi sekwencjami DNA, umożliwiając jakościową i ilościową analizę SNP poprzez detekcję sygnału. W porównaniu z tradycyjną metodą diagnostyczną pojedynczej komórki, mikromacierz SNP jest metodą o wysokiej przepustowości, która jest w stanie przeprowadzić tysiące reakcji na powierzchni chipa oligonukleotydowego w jednym czasie.
Przebieg pracy mikromacierzy SNP
Ogólny przebieg pracy mikromacierzy SNP przedstawiono na rycinie 3. W skrócie można powiedzieć, że istnieje kilka procesów: SNP chip fabrication, sample genomic DNA preparation, hybridization, and fluorescence scanning.
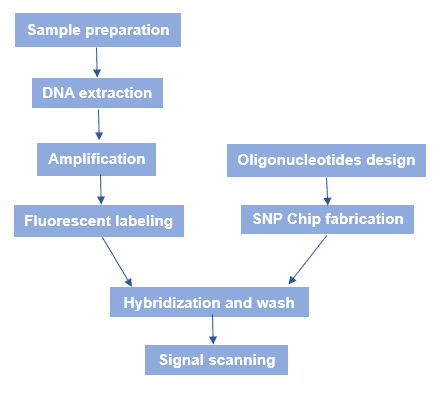 Figure 3. Przepływ pracy mikromacierzy SNP.
Figure 3. Przepływ pracy mikromacierzy SNP.
- Wytwarzanie chipów SNP
Zaprojektowane wcześniej chipy oligonukleotydowe lub cDNA są ułożone w sposób uporządkowany i w dużej gęstości na szklanym nośniku w celu wykonania mikromacierzy lub chipów rdzeniowych. Metody wytwarzania chipów obejmują światłowodową syntezę in situ, metodę rozpylania chemicznego, metodę kontaktowego powlekania kropek, syntezę kontrolowaną DNA in situ, metodę bezkontaktowego drukowania mikromechanicznego TOPSPOT® i odtwarzanie miękką litografią itp. Na chipie o powierzchni 1 cm2 można obecnie umieścić łącznie 4×105 różnych cząsteczek DNA. Technologia Affymetrix GeneChip® umożliwia syntezę oligonukleotydów o wysokiej gęstości, in situ, i była pionierem w tej dziedzinie. - Przygotowanie genomowego DNA próbki
W pierwszej kolejności przygotowuje się materiał próbki. Następnie genomowe DNA jest ekstrahowane i amplifikowane metodą PCR. Wreszcie, do jego znakowania stosuje się różne barwniki fluorescencyjne. - Hybrydyzacja i skanowanie
Genomowe DNA znakowane fluorescencyjnie jest hybrydyzowane z mikromacierzą SNP w odpowiednich warunkach reakcji. Po umyciu, specyficzny skaner fluorescencyjny jest używany do skanowania fluorescencji. Następnie komputerowa analiza obrazu jest wykonywana w celu przetworzenia danych przed wykonaniem analizy bioinformatycznej genu docelowego.
W CD Genomics zajmujemy się dostarczaniem niezawodnych usług genotypowania SNP, w tym genotypowania poprzez sekwencjonowanie, mikromacierze SNP, genotypowanie SNP MassARRAY, Hi-SNPseq, SNaPshot Multiplex System do genotypowania SNP i genotypowanie SNP TaqMan.
- Iwamoto K, Bundo M, Ueda J, et al. Detection of chromosomal structural alterations in single cells by SNP arrays: a systematic survey of amplification bias and optimized workflow. Plos One, 2007, 2(12): e1306.
- Ho C C, Mun K S, Naidu R. SNP array technology: an array of hope in breast cancer research. Malaysian Journal of Pathology, 2013, 35(1):33-43.
- Ahmad A, Iqbal M A. Significance of genome-wide analysis of copy number alterations and UPD in myelodysplastic syndromes using combined CGH-SNP arrays. Current Medicinal Chemistry, 2012, 19(22).
- Sund K L, Zimmerman S L, Thomas C, et al. Regions of homozygosity identified by SNP microarray analysis aid in the diagnosis of autosomal recessive disease and incidentally detect parental blood relationships. Genetics in Medicine Official Journal of the American College of Medical Genetics, 2013, 15(1):70-78.
- Liu S, Zhou Z, Lu J, et al. Generation of genome-scale gene-associated SNPs in catfish for the construction of a high-density SNP array. Bmc Genomics, 2011, 12(1):53.