Dada a relativa simplicidade estrutural e repetibilidade do DNA, seguiria que proteínas que se ligam especificamente a ele poderiam ter motivos de domínio de ligação de DNA comum, mas com cadeias laterais específicas de aminoácidos permitindo interações de ligação específicas.
- helix-turn-helix: encontrado nas proteínas de ligação do DNA procariótico.
Figure: helix-turn-helix
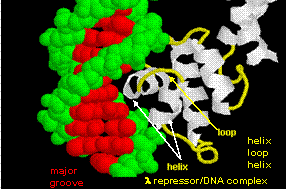
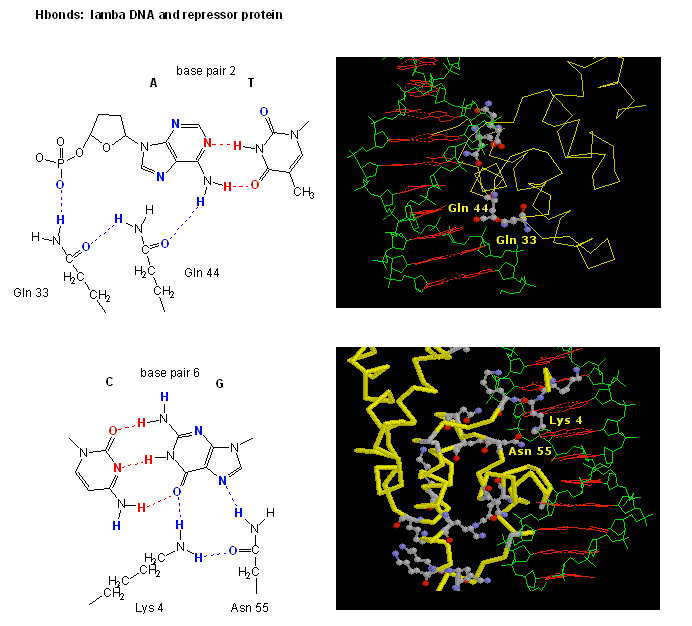
Os números mostram duas dessas proteínas, o cro repressor da bacteriófago 434 e o lambda repressor da bacteriófago lambda. (Os bacteriófagos são vírus que infectam a bactéria bacteia.) Observe como a especificidade é alcançada, em parte, pela formação de ligações H específicas entre a proteína e o maior bosque do DNA do operador.
Figure: Lambda Repressor/DNA Complexo
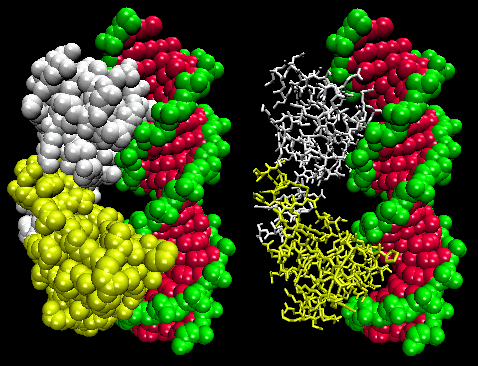
Figure: Interações H Bond betweenλ repressor e DNA
![]() Jmol: Atualizado Repressor Lambda/DNA complexo Jmol14 (Java) | JSMol (HTML5)
Jmol: Atualizado Repressor Lambda/DNA complexo Jmol14 (Java) | JSMol (HTML5)
- dedo de zinco: (eukaryotes) Estas proteínas têm um motivo de sequência comum de X3-Cys-X2-4-Cys-X12-His-X3-4-His-X4- no qual X é qualquer aminoácido. Zn2+ é tetrahedralmente coordenado com os Cys e Suas cadeias laterais, que estão em um dos dois fios beta antiparalelos, e uma hélice alfa, respectivamente. O dedo de zinco, estabilizado pelo zinco, liga-se à ranhura principal do ADN. O dedo de zinco, estabilizado pelo zinco, liga-se à ranhura principal do DNA: Atualizado Zif268:DNA Complex Jmol14 (Java) | JSMol (HTML5)
Zn proteínas do dedo, das quais 900 são codificadas no genoma humano (incluindo a proteína de ligação dos isoladores eucarióticos CTCF descrita acima) pode ser mobilizada para reparar mutações específicas em células, que se realizadas em uma porcentagem suficientemente alta de células mutantes poderiam curar doenças genéticas específicas, tais como algumas formas de doença imunodeficiente combinada grave. Nesta nova técnica (Urnov et al, 2005), os domínios de ligação de múltiplos dedos Zn ligados (um dos naturais ou formas mutantes produzidas em laboratório), cada um específico para uma determinada sequência de nucleotídeos, está ligado a uma endonuclease não específica, derivada da enzima FokI. A nuclease está ativa na forma dimérica, portanto o complexo ativo requer dois domínios endonuclease, cada um ligado a quatro domínios Zn de dedos diferentes, para ser montado no local alvo. A especificidade da ligação é alcançada através da selecção pelos domínios dos dedos Zn. Um nick é então feito pelo DNA pelo nuclease, e os mecanismos de reparação da célula hospedeira seguem. Este processo envolve a separação dos fios, a recombinação homóloga da região nick com DNA complementar dentro da célula, e a reparação do nick. Se o excesso de ADN do tipo selvagem (não mutado) for adicionado às células e usado como modelo, a mutação normal de reparação do ADN fixaria a mutação. Urnov et al mostraram que até 20% das células cultivadas que contêm uma mutação podem ser reparadas no laboratório. Se essas células ganhassem uma vantagem de crescimento seletivo, as células mutantes seriam eventualmente substituídas por células do tipo selvagem.
- receptores hormonais de esteróides: (eucariotas) Em contraste com a maioria dos hormônios, que se ligam aos receptores de superfície celular, os hormônios esteróides (derivados do colesterol) passam através da membrana celular e se ligam aos receptores citoplasmáticos através de um domínio de ligação hormonal. Isto muda a forma do receptor que depois se liga a um local específico no DNA (elemento de resposta hormonal) através de um domínio de ligação ao DNA. Em uma estrutura análoga ao dedo de zinco, Zn 2+ é tetrahedralmente coordenado a 4 Cys, em uma estrutura globular que se liga como um dímero a duas sequências idênticas, mas invertidas de DNA (palíndromo) dentro do bosque principal. (Exemplos de palíndromos: “Able was I ere I saw Elba; Dennis e Edna dine, said I, as Enid and Edna sinned.
Considerar o receptor glucocorticoide (GR) como um exemplo específico. Ele liga o DNA como um dímero. Os dois domínios de ligação de DNA do dímero associam-se com dois sulcos principais adjacentes do DNA na sequência de ligação da GR (GBS), uma sequência curta de DNA dentro do promotor. Meijsing, et al. descobriram que o GBS não só age como um local de ligação da GR, permitindo a transcrição de genes, mas também afeta a conformação do receptor, fazendo com que a transcrição de genes seja regulada de outra forma. O grupo construiu “genes repórteres” da luciferase que têm GBS ligados ao gene para a proteína luciferase, que expressaria a proteína luciferase (que fluoresce) se eles estivessem sendo transcritos, com a GBS. Descobriram que a atividade transcripcional relativa não estava correlacionada com a afinidade relativa de ligação da GR à SBC. Os PBGE que eram muito mais ativos que outros vinculados de forma comparável com os de menor atividade, enquanto os PBGE com atividade transcritiva semelhante vinculados com afinidades diferentes. Isto mostra que o PBE está conferindo uma função única à EM associada a ele (ou seja, a transcrição não é simplesmente afetada pelo fato de a EM estar ou não vinculada ao PBE). Um �lever arm� do receptor sofreu alterações conformacionais quando vinculado ao DNA, com alterações específicas da seqüência à qual estava vinculado. Uma proteína mutante, GR-γ, foi feita para ser idêntica à proteína do tipo selvagem, GR-α, exceto no braço de alavanca, foi constatado que tinham atividade transcripcional diferente, mesmo que estivessem ligados ao mesmo local no DNA, mostrando que o braço de alavanca e sua conformação afeta a transcrição.
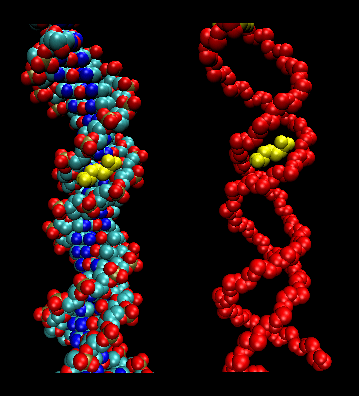
- fechos de correr (ou tesouras) leucina: (eucariotas) Estas proteínas contêm estiramentos de 35 aminoácidos nos quais o Leu é encontrado repetidamente em intervalos de 7 aminoácidos. Estas regiões da proteína formam hélices anfifílicas, com Leu em uma face, um Leu após duas voltas de uma hélice. Duas destas proteínas podem formar um dímero, estabilizado pela ligação destas hélices anfifílicas não polares e ricas em leucina uma à outra, formando uma bobina, tal como na miosina da proteína muscular. O zíper leucina representa o domínio de ligação da proteína. O domínio de ligação do DNA é encontrado nos primeiros 30 aminoácidos terminais N, que são básicos e formam uma hélice alfa quando a proteína se liga ao DNA. O zíper leucino então funciona para reunir duas proteínas de ligação de DNA, permitindo que as bases N-terminais interajam com o maior bosque de DNA de uma forma base-específica. Valina e isoleucina, juntamente com a leucina, são frequentemente encontradas em extensões de aminoácidos que podem interagir para formar outros tipos de bobinas enroladas.
Figure: fecho de correr leucina (feito com VMD)
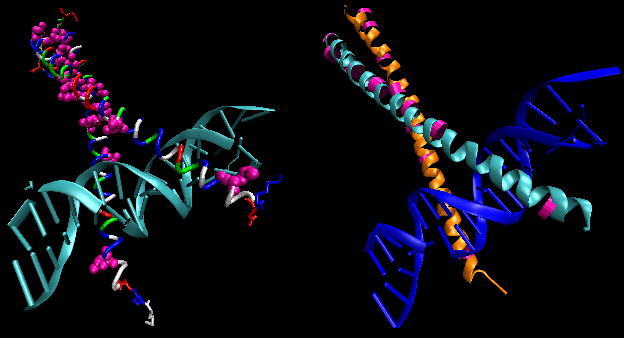
 Jmol: Atualizado Leucine Zipper Jmol14 (Java) | JSMol (HTML5)
Jmol: Atualizado Leucine Zipper Jmol14 (Java) | JSMol (HTML5) >
Assim como as nucleases dos dedos de zinco foram usadas para induzir o reparo de mutações, outro estudo do genoma do rato usado ZFNs especialmente projetados para causar quebras no ds-DNA que contém mutações do mecanismo de reparo do DNA impreciso (por NHEJ) e, portanto, continha mutações específicas (Geurts, et al. 2009). Este processo, �knockout do gene,� impede a produção da proteína normalmente transcrita pelo gene alvo. Foram utilizados ZFNs de cinco e seis dedos para alcançar um alto nível de especificidade na ligação ao gene alvo para três proteínas diferentes: proteína fluorescente verde (GFP), imunoglobulina M (IgM) e Rab38. O knockout foi bem sucedido em 12% dos ratos testados; estes animais não tinham proteína do tipo selvagem e não tinham expressão. Os ZFNs foram suficientemente específicos para que não fossem observadas mutações em nenhum dos 20 locais não-alvo previstos. Este estudo apoia a viabilidade do controle de transcrição e expressão para o tratamento de doenças e a importância da ligação específica.
Vimos que dois fatores principais contribuem para o reconhecimento específico do DNA pelas proteínas; a formação de ligações de hidrogênio para doadores e aceitadores de nucleotídeos específicos no sulco principal, e deformações dependentes da seqüência da hélice de DNA para formas alteradas com o aumento da afinidade dos ligantes protéicos. Por exemplo, a proteína Tata Binding Protein (TBP) pode interagir com um sulco menor ampliado na caixa TATA. Novas descobertas suportam que, além disso, as proteínas são capazes de utilizar informações em sulcos menores que se tornaram “estreitos” dependendo da sequência de nucleotídeos.
Tracks of DNA enriched in A can lead to twisting conformations that cause inter-base-pair hydrogen bonding in the major grooves, resulta no estreitamento dos sulcos menores. Altas quantidades de pares de bases AT estão concentradas em sulcos menores estreitos (largura <5.0 �) e os pares de bases CG são encontrados mais frequentemente em sulcos menores largos.
Como o estreitamento dos sulcos menores afeta o reconhecimento do DNA? Os sulcos menores estreitos aumentam o potencial eletrostático negativo do DNA, tornando-o um local mais específico e reconhecível. Os fosfatos da espinha dorsal do DNA estão mais próximos do meio da ranhura quando esta é estreita, correlacionando assim ranhuras menores estreitas com um potencial eletrostático mais negativo.
As partes menores das proteínas que interagem com o sulco contêm arginina cuja cadeia lateral pode ser acomodada no sulco menor mais estreito e negativo. As argininas podem ligar-se e, em alguns casos, inserir-se como motivos de sequência curta que melhoram a especificidade do reconhecimento da forma do ADN. O Arg é preferido ao Lys já que os raios efetivos da carga em Arg são maiores do que os do portador de carga em Lys. Isto levaria a uma diminuição da energia de desolação para Arg, o que promoveria a sua ligação ao bosque principal estreito. Esta descoberta mostra que “o papel da forma do DNA deve ser levado em consideração ao anotar todo o genoma e prever locais de ligação do fator de transcrição”.
Figure: Argumento em T3c Transposase binding in Narrowed Minor Grove of T3c Transposon
- Modelo deslizante para interação proteína/DNA
Contribuidores e Atribuições
- Prof. Henry Jakubowski (College of St. Benedict/St. John’s University)