Single nucleotide polymorphism
Single nucleotide polymorphism (SNP) refere-se à diferença de um único nucleotídeo na mesma posição na sequência de DNA genómico. Em geral, uma variação de um único nucleotídeo com uma frequência superior a 1% é chamada de SNP. Os SNPs envolvem apenas uma única variação de base, que pode ser causada por uma única transição ou transversalidade de base. Há aproximadamente um SNP por 1000 bases no genoma humano, e o número total de SNPs no genoma humano é de cerca de 3 x 106,
SNP tornou-se a terceira geração de marcadores genéticos. As características dos SNPs os tornam mais adequados para estudos sobre pleiotropia genética em características e doenças complexas, e reconhecimento genético baseado na população do que outros marcadores polimórficos. Os SNPs têm uma importância significativa na pesquisa biomédica. Os SNPs têm sido utilizados em estudos de associação de genomas como marcadores de alta resolução no mapeamento genético relacionado a doenças ou traços normais. Os SNPs sem um impacto observável no fenótipo (chamado mutações silenciosas) também podem ser úteis devido à sua quantidade e herança estável ao longo de gerações.
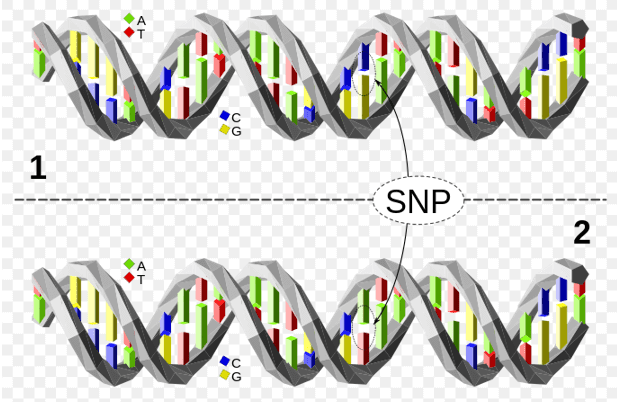 Figure 1. A molécula de DNA superior difere da molécula de DNA inferior em um único par de base (um polimorfismo C/A).
Figure 1. A molécula de DNA superior difere da molécula de DNA inferior em um único par de base (um polimorfismo C/A).
Microarray
O microarray de DNA refere-se a um chip genético com um grande número de seqüências de DNA de sonda em um arranjo específico imobilizado em um substrato sólido. O princípio é a teoria da hibridação do ácido nucleico, e a amostra de ADN detectada é hibridizada com o microarray de ADN e estendida. Subsequentemente, os fragmentos da reacção de ligação não suplementar no chip são lavados e o chip genético é sujeito a varrimento confocal a laser. As intensidades de sinal de fluorescência são então medidas e interpretadas como a abundância de diferentes genes através do processamento de certos dados.
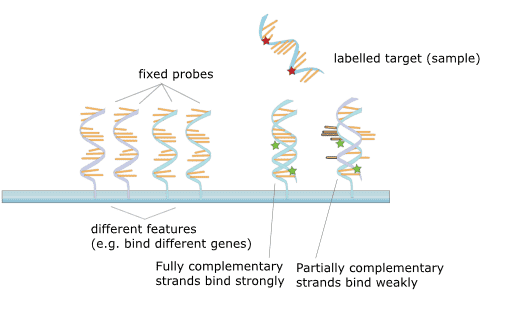 Figure 2. O princípio do microarray.
Figure 2. O princípio do microarray.
SNP microarray
SNP microarray utiliza sequências nucleotídicas conhecidas como sondas para hibridizar com as sequências de ADN testadas, permitindo a análise qualitativa e quantitativa do SNP através da detecção do sinal. Em comparação com o método tradicional de diagnóstico de célula única, o SNP microarray é um método de alto rendimento, capaz de realizar milhares de reações na superfície do chip oligonucleotídeo de uma vez.
O fluxo de trabalho do SNP microarray
O fluxo de trabalho geral do SNP microarray é mostrado na figura 3. Resumidamente falando, existem vários processos: Fabricação do chip SNP, preparação do DNA genômico da amostra, hibridação e varredura por fluorescência.
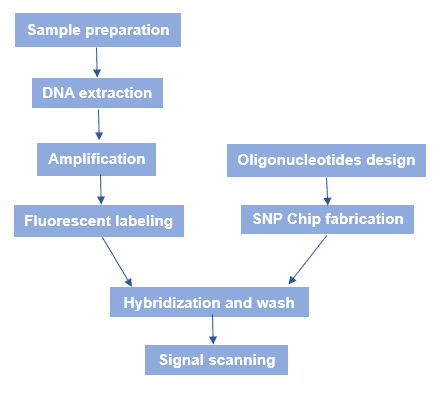 Figure 3. O fluxo de trabalho do SNP microarray.
Figure 3. O fluxo de trabalho do SNP microarray.
- Fabricação de chips SNP
Os chips de oligonucleotídeos ou cDNA pré-desenhados são dispostos ordenadamente e em alta densidade no suporte de vidro para fazer microarrays ou chips de núcleo. Os métodos de fabricação do chip incluem síntese in-situ guiada por luz, método de spray químico, método de revestimento por pontos de contato, síntese controlada por DNA in-situ, método de impressão micromecânica sem contato TOPSPOT® e reprodução litográfica suave, etc. Um total de 4×105 moléculas de DNA diferentes podem agora ser colocadas num chip de 1 cm2. A tecnologia Affymetrix GeneChip® permite a síntese de oligonucleotídeos de alta densidade in situ, e tem sido pioneira neste campo. - Preparação da amostra de DNA genômico
Primeiro, preparando o material da amostra. Em seguida, o DNA genômico é extraído e amplificado por PCR. Finalmente, diferentes corantes fluorescentes são usados para rotulá-lo. - Hibridação e varredura
O DNA genômico rotulado com fluorescência é hibridizado com microarray SNP sob condições de reação adequadas. Depois de lavado, um scanner de fluorescência específico é usado para digitalizar a fluorescência. Em seguida, a análise da imagem por computador é feita para processar os dados antes que a análise bioinformática do gene alvo seja realizada.
Na CD Genomics, estamos dedicados a fornecer serviços confiáveis de genotipagem SNP, incluindo genotipagem por sequenciamento, SNP microarray, MassARRAY SNP genotyping, Hi-SNPseq, SNaPshot Multiplex System for SNP genotyping, e TaqMan SNP genotyping.
- Iwamoto K, Bundo M, Ueda J, et al. Detecção de alterações estruturais cromossômicas em células únicas por matrizes SNP: um levantamento sistemático do viés de amplificação e fluxo de trabalho otimizado. Plos One, 2007, 2(12): e1306.
- Ho C C, Mun K S, Naidu R. SNP array technology: an array of hope in breast cancer research. Malaysian Journal of Pathology, 2013, 35(1):33-43.
- Ahmad A, Iqbal M A. Significância da análise de alterações no número de cópias e UPD em síndromes mielodisplásicas utilizando matrizes combinadas CGH-SNP. Current Medicinal Chemistry, 2012, 19(22).
- Sund K L, Zimmerman S L, Thomas C, et al. Regiões de homozigosidade identificadas pelo SNP microarray analysis ajudam no diagnóstico de doença autossômica recessiva e, incidentalmente, detectam relações sanguíneas parentais. Genetics in Medicine Official Journal of the American College of Medical Genetics, 2013, 15(1):70-78.
- Liu S, Zhou Z, Lu J, et al. Generation of genome-scale gene-associated SNPs in catfish for the construction of a high-density SNP array. Bmc Genomics, 2011, 12(1):53.