Primorfismul nucleotidic unic
Primorfismul nucleotidic unic (SNP) se referă la diferența de nucleotide unice în aceeași poziție în secvența ADN genomic. În general, o variație a unei singure nucleotide cu o frecvență mai mare de 1% se numește SNP. SNP-urile implică doar o singură variație a unei singure baze, care poate fi cauzată de o singură tranziție de bază sau de o singură transversiune. Există aproximativ un SNP la 1000 de baze în genomul uman, iar numărul total de SNP din genomul uman este de aproximativ 3 x 106.
SNP a devenit a treia generație de markeri genetici. Caracteristicile SNP-urilor fac ca acestea să fie mai potrivite pentru studii privind pleiotropia genetică în trăsături și boli complexe și pentru recunoașterea genetică la nivelul populației decât alți markeri polimorfici. SNP-urile au o importanță semnificativă în cercetarea biomedicală. SNP-urile au fost utilizate în studiile de asociere la nivelul întregului genom ca markeri de înaltă rezoluție în cartografierea genelor legate de boli sau trăsături normale. SNP-urile fără un impact observabil asupra fenotipului (numite mutații silențioase) pot fi, de asemenea, utile datorită cantității și moștenirii lor stabile de-a lungul generațiilor.
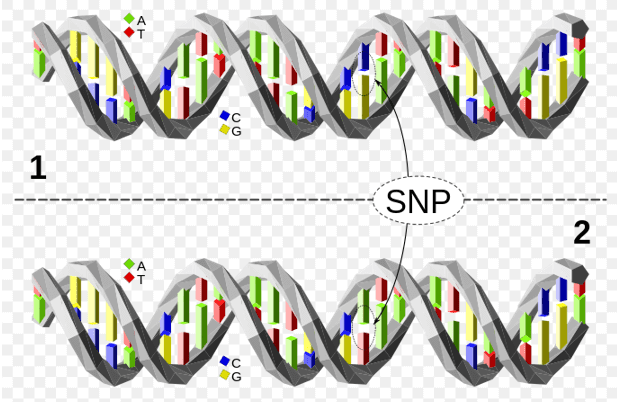 Figura 1. Molecula superioară de ADN diferă de molecula inferioară de ADN la o singură locație de pereche de baze (un polimorfism C/A).
Figura 1. Molecula superioară de ADN diferă de molecula inferioară de ADN la o singură locație de pereche de baze (un polimorfism C/A).
Microarray
Microarray-ul de ADN se referă la un cip genetic cu un număr mare de secvențe de ADN sondă într-un aranjament specific imobilizate pe un substrat solid. Principiul este teoria hibridizării acizilor nucleici, iar proba de ADN detectată este hibridizată cu microarray-ul de ADN și extinsă. Ulterior, fragmentele din reacția de legare necomplementară de pe cip sunt spălate, iar cipul genetic este supus scanării confocale cu laser. Intensitățile semnalului de fluorescență sunt apoi măsurate și interpretate ca abundență a diferitelor gene prin anumite prelucrări de date.
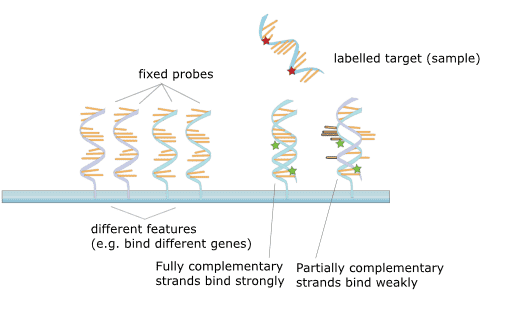 Figura 2. Principiul microarray.
Figura 2. Principiul microarray.
Microarray SNP
Microarray SNP utilizează secvențe de nucleotide cunoscute ca sonde pentru a se hibridiza cu secvențele de ADN testate, permițând analiza calitativă și cantitativă a SNP prin detectarea semnalului. În comparație cu metoda tradițională de diagnosticare cu o singură celulă, SNP microarray este o metodă de mare randament, care este capabilă să efectueze mii de reacții pe suprafața cipului oligonucleotidic la un moment dat.
Fluxul de lucru al SNP microarray
Fluxul de lucru general al SNP microarray este prezentat în figura 3. Pe scurt, există mai multe procese: Fabricarea cipului SNP, pregătirea ADN-ului genomic al probei, hibridizarea și scanarea prin fluorescență.
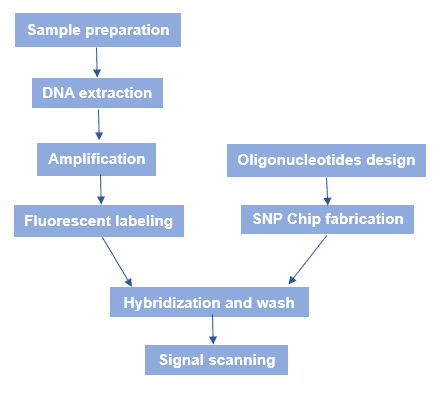 Figura 3. Fluxul de lucru al microrețelelor SNP.
Figura 3. Fluxul de lucru al microrețelelor SNP.
- Fabricarea cipurilor SNP
Pietricele de oligonucleotide sau cADN preproiectate sunt aranjate ordonat și în densitate mare pe suportul de sticlă pentru a realiza microrețele sau cipuri de bază. Metodele de fabricare a cipului includ sinteza in-situ ghidată de lumină, metoda de pulverizare chimică, metoda de acoperire cu puncte de contact, sinteza controlată in-situ a ADN-ului, metoda de imprimare micromecanică fără contact TOPSPOT® și reproducerea prin litografie moale etc. În total, pe un cip de 1 cm2 pot fi plasate acum 4 x 105 molecule diferite de ADN. Tehnologia Affymetrix GeneChip® permite sinteza de oligonucleotide de înaltă densitate, in situ, și a fost un pionier în acest domeniu. - Pregătirea probei de ADN genomic
În primul rând, pregătirea materialului de probă. Apoi se extrage ADN genomic și se amplifică prin PCR. În cele din urmă, se folosesc diferiți coloranți fluorescenți pentru a-l marca. - Hibridizare și scanare
ADN-ul genomic marcat cu fluorescență este hibridizat cu microarray-ul SNP în condiții de reacție adecvate. După ce se spală, se utilizează un scaner de fluorescență specific pentru a scana fluorescența. Apoi se face analiza imaginii computerizate pentru a procesa datele înainte de a se efectua analiza bioinformatică a genei țintă.
La CD Genomics, suntem dedicați furnizării de servicii fiabile de genotipare SNP, inclusiv genotiparea prin secvențiere, microarray SNP, genotiparea SNP MassARRAY, Hi-SNPseq, SNaPshot Multiplex System pentru genotiparea SNP și genotiparea SNP TaqMan.
- Iwamoto K, Bundo M, Ueda J, et al. Detection of chromosomal structural alterations in single cells by SNP arrays: a systematic survey of amplification bias and optimized workflow. Plos One, 2007, 2(12): e1306.
- Ho C C, Mun K S, Naidu R. SNP array technology: an array of hope in breast cancer research. Malaysian Journal of Pathology, 2013, 35(1): 33-43.
- Ahmad A, Iqbal M A. Significance of genome-wide analysis of copy number alterations and UPD in myelodysplastic syndromes using combined CGH-SNP arrays. Current Medicinal Chemistry, 2012, 19(22).
- Sund K L, Zimmerman S L, Thomas C, et al. Regions of homozygosity identified by SNP microarray analysis aid in the diagnosis of autosomal recessive disease and incidentally detect parental blood relationships. Genetics in Medicine Official Journal of the American College of Medical Genetics, 2013, 15(1):70-78.
- Liu S, Zhou Z, Lu J, et al. Generation of genome-scale gene-associated SNPs in catfish for the construction of a high-density SNP array. Bmc Genomics, 2011, 12(1):53.
.