Yksittäisnukleotidipolymorfismi
Yksittäisnukleotidipolymorfismilla (engl. Single nucleotide polymorphism, SNP) tarkoitetaan yksittäisten nukleotidien eroavaisuutta samassa sijainnissa perimän DNA:n sekvenssissä. Yleensä yksittäisen nukleotidin vaihtelua, jonka esiintymistiheys on yli 1 %, kutsutaan SNP:ksi. SNP:t sisältävät vain yhden emäksen vaihtelun, joka voi johtua yhden emäksen siirtymisestä tai transversiosta. Ihmisen genomissa on noin yksi SNP 1000 emästä kohti, ja SNP:iden kokonaismäärä ihmisen genomissa on noin 3 x 106.
SNP:stä on tullut kolmannen sukupolven geneettisiä markkereita. SNP:iden ominaisuuksien vuoksi ne soveltuvat muita polymorfisia merkkiaineita paremmin monimutkaisten ominaisuuksien ja sairauksien geneettisen pleiotropian tutkimiseen ja populaatiopohjaiseen geenien tunnistamiseen. SNP:illä on merkittävä merkitys biolääketieteellisessä tutkimuksessa. SNP:tä on käytetty genominlaajuisissa assosiaatiotutkimuksissa korkean resoluution markkereina sairauksiin tai normaaleihin ominaisuuksiin liittyvässä geenikartoituksessa. SNP:t, joilla ei ole havaittavaa vaikutusta fenotyyppiin (ns. hiljaiset mutaatiot), voivat myös olla hyödyllisiä niiden määrän ja sukupolvien yli tapahtuvan vakaan periytymisen vuoksi.
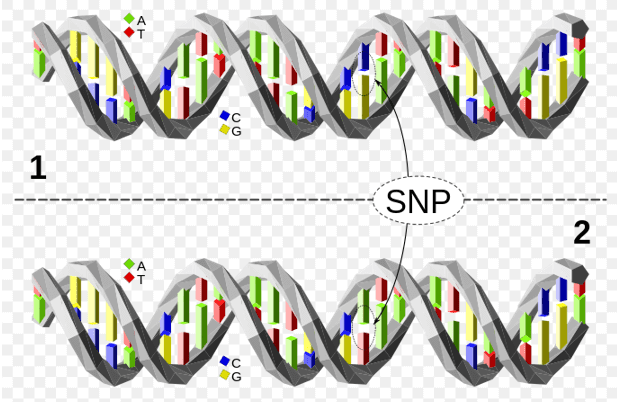 Kuva 1. SNP-mikrosirujen periaatteet ja työnkulku. Ylempi DNA-molekyyli eroaa alemmasta DNA-molekyylistä yhden emäsparin kohdalla (C/A-polymorfismi).
Kuva 1. SNP-mikrosirujen periaatteet ja työnkulku. Ylempi DNA-molekyyli eroaa alemmasta DNA-molekyylistä yhden emäsparin kohdalla (C/A-polymorfismi).
Mikrosiru
DNA-mikrosirulla tarkoitetaan geenisirua, jossa on suuri määrä koetin-DNA-sekvenssejä tietyssä järjestyksessä immobilisoituna kiinteälle substraatille. Periaate on nukleiinihappohybridisaatioteoria, ja havaittu näyte-DNA hybridisoidaan DNA-mikrosirun kanssa ja laajennetaan. Tämän jälkeen sirulla olevat ei-komplementaarisen sitoutumisreaktion fragmentit pestään pois, ja geenisiru altistetaan laser-konfokaaliselle skannaukselle. Tämän jälkeen mitataan fluoresenssisignaalin voimakkuudet, ja ne tulkitaan eri geenien runsaudeksi tietyn tietojenkäsittelyn avulla.
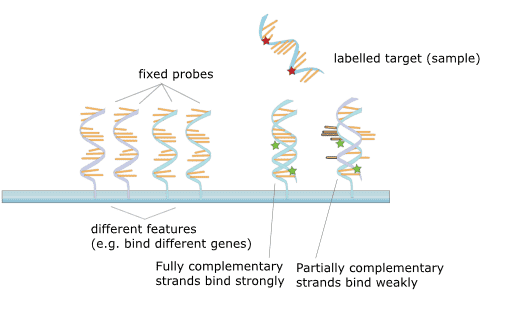 Kuva 2. SNP-mikrosirujen periaatteet ja työnkulku. Mikrosirujen periaate.
Kuva 2. SNP-mikrosirujen periaatteet ja työnkulku. Mikrosirujen periaate.
SNP-mikrosiru
SNP-mikrosiru käyttää tunnettuja nukleotidisekvenssejä koettimina, jotka hybridisoituvat testattavien DNA-sekvenssien kanssa, mikä mahdollistaa kvalitatiivisen ja kvantitatiivisen SNP-analyysin signaalin havaitsemisen avulla. Perinteiseen yksittäisen solun diagnostiikkamenetelmään verrattuna SNP-mikrosiru on korkean läpimenon menetelmä, joka pystyy suorittamaan tuhansia reaktioita oligonukleotidisirun pinnalla kerralla.
SNP-mikrosirun työnkulku
SNP-mikrosirun yleinen työnkulku on esitetty kuvassa 3. Lyhyesti sanottuna prosesseja on useita: SNP-sirujen valmistus, näytteen genomisen DNA:n valmistelu, hybridisointi ja fluoresenssiskannaus.
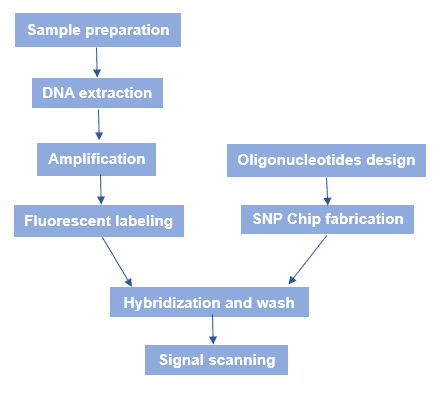 Kuvio 3. SNP-mikrosirujen valmistus. SNP-mikrosirujen työnkulku.
Kuvio 3. SNP-mikrosirujen valmistus. SNP-mikrosirujen työnkulku.
- SNP-sirujen valmistus
Ensisijaisesti suunnitellut oligonukleotidi- tai cDNA-sirut järjestetään järjestyksessä ja suurella tiheydellä lasikannattimelle mikrosirujen tai ydinsirujen valmistamiseksi. Sirun valmistusmenetelmiin kuuluvat valo-ohjattu in situ -synteesi, kemiallinen suihkutusmenetelmä, kontaktipistepinnoitusmenetelmä, in situ DNA-ohjattu synteesi, kosketukseton mikromekaaninen tulostusmenetelmä TOPSPOT® ja pehmeä litografinen jäljentäminen jne. Yhteensä 4×105 erilaista DNA-molekyyliä voidaan nyt sijoittaa 1 cm2 kokoiselle sirulle. Affymetrix GeneChip® -teknologia mahdollistaa korkean tiheyden in situ -oligonukleotidisynteesin, ja se on ollut edelläkävijä tällä alalla. - Näytteen genomisen DNA:n valmistelu
Ensin valmistetaan näytemateriaali. Sitten genominen DNA uutetaan ja monistetaan PCR:llä. Lopuksi sen merkitsemiseen käytetään erilaisia fluoresoivia väriaineita. - Hybridisointi ja skannaus
Fluoresenssilla merkitty genominen DNA hybridisoidaan SNP-mikrosirujen kanssa sopivissa reaktio-olosuhteissa. Pesun jälkeen käytetään spesifistä fluoresenssiskanneria fluoresenssin skannaamiseen. Sitten tehdään tietokonekuva-analyysi datan käsittelemiseksi, ennen kuin suoritetaan kohdegeenin bioinformatiikan analyysi.
Me CD Genomicsilla olemme omistautuneet tarjoamaan luotettavia SNP-genotyypin määrityspalveluja, mukaan lukien genotyypin määritys sekvensoinnilla, SNP-mikrosirulla, MassARRAY SNP-genotyypin määrityksessä, Hi-SNPseq:lla, SNaPshot-multiplex-järjestelmällä SNP-genotyypin määritystä varten ja TaqMan SNP-genotyypin määrityksellä.
- Iwamoto K, Bundo M, Ueda J, et al. Detection of chromosomal structural alterations in single cells by SNP arrays: a systematic survey of amplification bias and optimized workflow. Plos One, 2007, 2(12): e1306.
- Ho C C, Mun K S, Naidu R. SNP-matriisitekniikka: toivon joukko rintasyövän tutkimuksessa. Malaysian Journal of Pathology, 2013, 35(1):33-43.
- Ahmad A, Iqbal M A. Significance of genome-wide analysis of copy number alterations and UPD in myelodysplastic syndromes using combined CGH-SNP arrays. Current Medicinal Chemistry, 2012, 19(22).
- Sund K L, Zimmerman S L, Thomas C, et al. Regions of homozygosity identified by SNP microarray analysis help in the diagnosis of autosomal recessive disease and incidentally detect parental blood relationships. Genetics in Medicine Official Journal of the American College of Medical Genetics, 2013, 15(1):70-78.
- Liu S, Zhou Z, Lu J, et al. Generation of genome-scale gene-associated SNPs in catfish for the construction of a high-density SNP array. Bmc Genomics, 2011, 12(1):53.