Editorial om forskningsämnet
The Role of AAA+ Proteins in Reparation och nedbrytning av proteiner
ATPaser som är associerade med olika cellulära aktiviteter (AAA+) utgör en superfamilj av proteiner som utför ett stort antal olika funktioner som är viktiga för cellfysiologin, Detta inkluderar kontroll av proteinhomeostas, DNA-replikation, rekombination, kromatinremodellering, bearbetning av ribosomalt RNA, molekylär målinriktning, organell biogenes och membranfusion (Hanson och Whiteheart, 2005); Erzberger och Berger, 2006; Snider et al., 2008). Medlemmar av denna superfamilj definieras av närvaron av en så kallad AAA+-domän som innehåller de kanoniska Walker A- och B-motiven som krävs för ATP-bindning och hydrolys (Hanson och Whiteheart, 2005). Typiskt sett kodar genomerna för cirka tio till flera hundra AAA+-familjemedlemmar (tabell 1; Finn et al., 2017), varav var och en tros vara anpassad till specifika funktionella nischer som kräver exakta mekanismer för substratigenkänning och -bearbetning (Hanson och Whiteheart, 2005). AAA+-proteinernas slående adaptiva utstrålning för att fungera i olika miljöer illustrerar AAA+-domänens mångsidiga användbarhet (Erzberger och Berger, 2006). AAA+-proteiner bildar vanligtvis hexameriska komplex och fungerar som motorer för att omforma andra proteiner, DNA/RNA eller multikomponentkomplex (figur 1). Faktum är att många chaperoner och ATP-beroende proteaser är eller har underenheter som tillhör denna superfamilj (figur 1; Olivares et al., 2016).
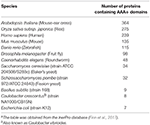
Tabell 1. Antal AAA+ proteiner i modellorganismera.
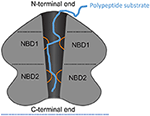
Figur 1. Schematisk bild av ClpA som ett exempel på AAA+ hexamer. Schematisk representation av domänarkitekturen och interaktionerna hos ClpA som ett exempel på AAA+ hexamer med två AAA+-domäner per monomer. Här visas en utklippt sidovy av ClpA-hexamern för att skildra den centrala, polypeptidledande kanalen. ClpA innehåller tre domäner, inklusive en N-terminal domän och två AAA+-domäner: nukleotidbindande domän 1 och 2 (NBD1 och NBD2). Den N-terminala domänen interagerar med regulatorer av substratspecificitet, medan den C-terminala änden interagerar med kammarproteaset ClpP. Mobila slingor från NBD1 och NBD2 (orange) projicerar in i den centrala kanalen och engagerar polypeptidsubstratet (blått) och möjliggör därmed koppling av ATP-hydrolys till polypeptidtranslokation över den centrala kanalen.
Under de senaste åren har det gjorts betydande framsteg när det gäller att identifiera strukturen och den funktionella mekanismen hos ett stort antal AAA+-proteiner (Gates et al., 2017; Puchades et al., 2017; Ripstein et al., 2017; Zehr et al., 2017). I detta forskningsämne förmedlas flera delar av dessa spännande framsteg i 21 artiklar, som omfattar en detaljerad strukturell och mekanistisk bild av flera AAA+ chaperoner och proteaser, t.ex: ClpX (Alhuwaider och Dougan; Bittner et al.; Elsholz et al.; LaBreck et al.; Vass et al.), ClpA (Bittner et al.; Duran et al.), ClpB och Hsp104 (Chang et al.; Duran et al.; Franke et al.; Johnston et al.), Hsp78 (Abrahão et al.), ClpC (Alhuwaider och Dougan; Elsholz et al.), ClpE (Elsholz et al.), Pontin (Mao och Houry), Reptin (Mao och Houry), FtsH (Alhuwaider och Dougan), 19S-proteasomen (Snoberger et al.; Yedidi et al.), Lon (Alhuwaider och Dougan; Bittner et al.; Fishovitz et al.), p97 (Hänzelmann och Schindelin; Saffert et al.; Ye et al.), Pex1/6 (Saffert et al.), CbbQ (Mueller-Cajar), rubiscoaktivas (Bhat et al.), torsiner (Chase et al.) och mitokondriella AAA+ proteaser (Glynn). Här presenterar vi dessa fascinerande arbeten.
Studies on ClpXP, Lon, and Related ATP-Dependent Proteases
I sin forskningsartikel ”The Protein Chaperone ClpX Targets Native and Non-native Aggregated Substrates for Remodeling, Disassembly, and Degradation with ClpP”, LaBreck et al. en rad eleganta experiment för att fastställa att ClpX har disaggregationsaktivitet mot polypeptider som innehåller specifika ClpX-igenkänningssignaler (LaBreck et al.). I närvaro av ClpP kopplar ClpX disaggregering av dessa substrat till deras nedbrytning. Viktigt är att de också fastställer att ClpXP förhindrar ackumulering av aggregat som bildas av proteiner med ClpX-igenkänningssignaler in vivo (LaBreck et al.). Dessa studier belyser ClpX som ett proteindisaggregas, vilket tidigare var undervärderat.
I sin forskningsartikel ”The Essential Role of ClpXP in Caulobacter crescentus Requires Species Constrained Substrate Specificity” utforskar Vass et al. artspecifika funktioner hos ClpX (Vass et al.). Märkligt nog är ClpX viktigt för vissa arter, t.ex. C. crescentus, men inte för andra bakterier, t.ex. E. coli (Vass et al.). Viktigt är att E. coli ClpX inte kunde komplettera C. crescentus ClpX in vivo (Vass et al.). Denna brist på aktivitet berodde på artspecifika skillnader i ClpX:s N-terminala domän, som är kritisk för att bearbeta replikationsklampans laddningsunderenhet DnaX i C. crescentus. Således kan små skillnader i ClpX-specificitet vara särskilt kritiska för specifika bakteriearter.
I sin översikt om ”Functional Diversity of AAA+ Protease Complexes in Bacillus subtilis” diskuterar Elsholz et al. funktionerna hos flera AAA+ proteaser i B. subtilis, dvs: ClpCP, ClpEP, ClpXP, ClpYQ, LonA/B och FtsH (Elsholz et al.). De diskuterar hur olika stressreaktioner styr deras uttryck och de fenotyper som observeras vid borttagning av dessa olika proteaser. Vissa av dessa proteasers förmåga att kontrollera kompetens, sporulering, motilitet och biofilmsbildning beskrivs. Slutligen diskuterar författarna inriktning på dessa proteaser för utveckling av nya antibiotika.
I sin översikt med titeln ”AAA+ Machines of Protein Destruction in Mycobacteria” diskuterar Alhuwaider et al. (Alhuwaider och Dougan) de senaste framstegen när det gäller att bestämma strukturen och funktionen hos AAA+-proteaser i mykobakterier. Dessa proteaser är: ClpXP1P2, ClpC1P1P2, Lon, FtsH och Mpa. Författarna diskuterar också Pup-proteasomsystemet (PPS) som finns i mykobakterier och som motsvarar ubiquitin-proteasomsystemet i eukaryoter. Alhuwaider et al. avslutar sedan med en diskussion om nya föreningar som dysregulerar eller hämmar aktiviteten hos ClpP1P2 och andra som dysregulerar ClpC1. Dessa föreningar har lovande aktiviteter mot mykobakterier.
I en forskningsartikel med titeln ”The Copper Efflux Regulator CueR Is Subject to ATP-Dependent Proteolysis in Escherichia coli” visar Bittner et al. att AAA+-proteaserna Lon, ClpXP och ClpAP ansvarar för nedbrytningen av E. coli CueR, som är en transkriptionsfaktor som kontrollerar induktionen av kopparutflödet Cue-systemet (Bittner et al.). Författarna konstaterade att för att CueR ska kunna kännas igen av AAA+-proteaserna krävs CueR:s tillgängliga C-terminus. De drar slutsatsen att ATP-beroende proteaser krävs för kopparhomeostas i E. coli.
Fishovitz et al. genomför en detaljerad jämförelse mellan mänsklig och E. coli Lon i sin forskningsartikel med titeln ”Utilization of Mechanistic Enzymology to Evaluate the Significance of ADP Binding to Human Lon Protease” (Fishovitz et al.). Genom att använda en detaljerad mekanistisk studie fann de att till skillnad från E. coli Lon har humant Lon låg affinitet för ADP trots att det uppvisar jämförbara kcat- och KM-värden i ATPaseaktiviteten. De föreslår att mänsklig Lon inte regleras av en substratfrämjande ADP/ATP-utbytesmekanism. Dessa skillnader mellan mänsklig och E. coli Lon kan möjliggöra framtida utveckling av artspecifika Lon-hämmare.
I sin översikt om ”Multifunctional Mitochondrial AAA Proteases” diskuterar Dr. Glynn de två mitokondriella AAA-proteaserna, i-AAA och m-AAA (Glynn). Båda är mitokondriella proteiner i det inre membranet. I-AAA projicerar dock ATPas- och proteasdomänerna in i det mitokondriella intermembranutrymmet, medan m-AAA-proteasen projicerar de katalytiska domänerna in i matrisen. Strukturerna hos dessa proteaser diskuteras liksom deras funktionsmekanism. Proteaserna kan utföra fullständig substratnedbrytning, men kan också bara klyva vissa substrat, till exempel för MrpL32 och Atg32.
ClpB och Hsp104
I sin minirecension, ”Structural Elements Regulating AAA+ Protein Quality Control Machines”, Chang et al. diskuterar de hur porslinga-1, Inter-Subunit Signaling motivet och Pre-Sensor I insert motivet kan bidra till aktiviteten hos två Hsp100 disaggregaser, bakteriella ClpB och jäst Hsp104 (Chang et al.). De föreslår en modell för hur dessa strukturella element kan göra det möjligt att koppla AAA+ ATPase-cykeln till substrattranslokation över ClpB:s och Hsp104:s centrala kanal. Denna process av polypeptidtranslokation tros ligga till grund för hur ClpB och Hsp104 extraherar polypeptider från aggregerade strukturer (Chang et al.).
Duran et al. ger en ”Comparative Analysis of the Structure and Function of AAA+ Motors ClpA, ClpB, and Hsp104: Common Threads and Disparate Functions” i sin översikt (Duran et al.). De diskuterar förmågan hos dessa tre AAA+-proteiner (ClpA, ClpB och Hsp104) att transportera polypeptider genom sina hexameriska komplex. Alla dessa proteiner har två AAA+-domäner och är kända för att veckla ut proteiner. Det är viktigt att ClpB och Hsp104 också är kända för att fungera som disaggregaser, medan ClpA kan bilda ett komplex med ClpP-proteaset. Författarna lyfter fram behovet av att använda kinetiska metoder för övergående tillstånd för att undersöka de kinetiska mekanismerna hos dessa motorproteiner. De beskriver hur användningen av sådana metoder gjorde det möjligt för dem att visa att till exempel ClpA translokaliserar polypeptider vid cirka 20 aa s-1, medan ClpA i komplex med ClpP-proteaset har en ännu högre translokaliseringshastighet på cirka 35 aa s-1. Författarna diskuterar också betydelsen av chaperonet Hsp70 för funktionen av ClpB/Hsp104,och observationen av artspecificitet i interaktionen mellan Hsp70 och ClpB/Hsp104.
I sin forskningsartikel med titeln ”Mutant Analysis Reveals Allosteric Regulation of ClpB Disaggregase” genomför Franke et al. en mutationsanalys av E. coli ClpB disaggregase för att karakterisera dess allosteriska reglering (Franke et al.). ClpB kan delas in i en N-terminal domän och två AAA+-domäner som skiljs åt av en spiralformad region som kallas M-domän. Författarna identifierar en mycket konserverad rest i den första AAA+-domänen, A328. ClpB-A328V-mutanten visade sig ha mycket hög ATPasaktivitet och uppvisade celltoxicitet. Oväntat nog berodde den höga ATPasaktiviteten hos ClpB-A328V huvudsakligen på den andra AAA+-ringen, vilket bedömdes med hjälp av masspektrometri för amidväteutbyte. Författarna drar slutsatsen att A328 är en avgörande rest för att kontrollera ATP-hydrolysen i ClpB:s båda AAA+-ringar.
I sin forskningsartikel med titeln ”Substrate Discrimination by ClpB and Hsp104” beskriver Johnston et al. de medfödda substratpreferenserna hos ClpB och Hsp104 i avsaknad av chaperonsystemen DnaK och Hsp70 (Johnston et al.). De visar att substratspecificiteten bestäms av den första AAA+-domänen i varje protein. De kom fram till denna slutsats genom att testa de två chaperonerna med avseende på deras förmåga att verka på flera modellsubstrat. De testade också olika chimärer av de två chaperonerna.
I ”Hsp78 (78 kDa Heat Shock Protein), a Representative AAA Family Member Found in the Mitochondrial Matrix of Saccharomyces cerevisiae” diskuterar Abrahão et al. strukturen och funktionen hos Hsp78 (Abrahao et al.). Hsp78 är den mitokondriella paralogen till Hsp104, som fungerar vid disaggregering och reaktivering av proteiner (Abrahao et al.). Märkligt nog försvann Hsp104 och Hsp78 vid övergången från protozoer till metazoer (Abrahao et al.). Abrahao et al. diskuterar dock förekomsten av ANKCLP, som uppträder tillsammans med Hsp78 och Hsp104 i protozoer och överlever den evolutionära övergången till metazoer. ANKCLP har en AAA+-domän som liknar nukleotidbindande domän 2 (NBD2) hos Hsp104 och Hsp78, men är i övrigt mycket avvikande. Intressant nog orsakar mutationer i ANKCLP 3-metylglutakonsyrauri, progressiv hjärnatrofi, intellektuell funktionsnedsättning, medfödd neutropeni, grå starr och rörelsestörningar hos människor (Abrahao et al.).
p97
I sin översikt ”Structure and Function of p97 and Pex1/6 Type II AAA+ Complexes” diskuterar Saffert et al. två olika AAA+-komplex som remodellerar ubiquitinerade substratproteiner (Saffert et al.). En funktion av p97 är att förflytta ubiquitinerade substrat från ER-membranet till proteasomen under ER-associerad nedbrytning (Saffert et al.). Däremot är Pex1/Pex6 en heterohexamerisk motor som består av alternerande Pex1- och Pex6-underenheter och som är nödvändig för peroxisomernas biogenes och funktion. Nya cryo-elektronmikroskopiska (cryo-EM) strukturer av p97 och Pex1/6 diskuteras och viktiga strukturella skillnader lyfts fram.
I sin översikt med titeln ”A Mighty ’Protein Extractor’ of the Cell: Structure and Function of the p97/CDC48 ATPase” sammanfattar Ye et al. den nuvarande kunskapen om p97:s struktur och funktion och dess roll i flera sjukdomar (Ye et al.). p97 har två AAA+-domäner som är förbundna med en kort länk. Den har också en N-terminal domän som förmedlar dess interaktioner med olika adaptorproteiner. Författarna ger en detaljerad diskussion om p97:s struktur och effekten av nukleotider på dess olika konformationer. Studierna bygger på användning av tekniker som EM, röntgenkristallografi och atomkraftmikroskopi med hög hastighet. Författarna diskuterar sedan de multicellulära funktionerna hos detta mycket konserverade protein, inklusive dess roll i ER-associerad proteinnedbrytning (ERAD), mitokondrieassocierad nedbrytning (MAD) genom att extrahera polypeptider från mitokondriernas yttre membran och ribosomassocierad nedbrytning (RAD). Slutligen ger Ye et al. en sammanfattning av p97-mutationer som leder till flera mänskliga sjukdomar, t.ex. IBMPFD (Inclusion Body Myopathy associated with Paget’s disease of the bone and Frontotemporal Dementia)], FALS (familjär amyotrofisk lateralskleros), CMT2Y (Charcot-Marie-Tooth sjukdom, typ 2Y), hereditär spastisk paraplegi (HSP), Parkinsons sjukdom (PD) och Alzheimers sjukdom (AD).
I sin översikt om p97 med titeln ”The Interplay of Cofactor Interactions and Post-translational Modifications in the Regulation of the AAA+ ATPase p97” diskuterar Hänzelmann och Schindelin hur olika kofaktorer modulerar aktiviteten hos ATPaset p97 (Hanzelmann och Schindelin). De lyfter fram det faktum att p97:s förmåga att vara involverad i ett stort antal cellulära processer beror på det stora antalet kofaktorer som interagerar med detta protein. De har belyst tre olika klasser av p97-kofaktorer, nämligen: (i) substratrekryterande kofaktorer som UBA-UBX-proteiner och UFD1-NPL4, (ii) substratbearbetande kofaktorer som ubiquitin (E3)-ligaser och deubiquitinaser (DUBs) och (iii) reglerande kofaktorer som UBX-proteinerna, som kan sekretessbelägga eller återvinna p97-hexamerer. Författarna diskuterar också den roll som posttranslationella modifieringar spelar för p97:s aktivitet och för dess interaktioner med dess kofaktorer och substrat.
AAA+ Proteins of the Proteasome
I ”AAA-ATPases in Protein Degradation”, Yedidi et al. … en genomgång av aktiviteterna hos Rpt1, Rpt2, Rpt3, Rpt4, Rpt5 och Rpt6, som är AAA+ ATPaserna i den eukaryota proteasomen, samt några av deras bakteriella släktingar, t.ex. PAN, Mpa och VAT (Yedidi et al.). De fokuserar på ny teknik för att förstå hur dessa AAA+ ATPaser fungerar genom att translokera oveckade polypeptider till proteasets proteolytiska kammare (Yedidi et al.). Konformationsförändringar inom AAA+-ringen och proteaset med intilliggande kammare verkar generera en peristaltisk pumpmekanism för att leverera substrat för nedbrytning (Yedidi et al.).
I sin forskningsartikel ”The Proteasomal ATPases Use a Slow but Highly Processive Strategy to Unfold Proteins” skriver Snoberger et al. att proteasomala AAA+-proteiner använder en motorisk mekanism med låg hastighet men med hög processivitet för att leverera substrat till proteasomens proteolytiska kavitet (Snoberger et al.). Denna mekanism står i kontrast till ClpX, som utnyttjar en motorisk mekanism med hög hastighet men mindre processiv för att leverera substrat till ClpP-proteaset för nedbrytning. Dessa skillnader i motormekanism kan ha utvecklats som svar på olika krav från deras specifika klientel.
Rubisco Activases
I sin översikt om ”Rubisco Activases: AAA+ Chaperones Adapted to Enzyme Repair” diskuterar Bhat et al. den unika funktionen hos Rubisco activase (Rca) vid ombyggnad av Rubisco (Bhat et al.). Rca är en AAA+-chaperon som är mycket konserverad i fotosyntetiska organismer från bakterier till högre växter. Rubisco är ett enzym som heter Ribulose-1,5-bisfosfatkarboxylas/oxygenas och som är inblandat i fixeringen av koldioxid i atmosfären under fotosyntesen. Det är det mest förekommande proteinet på jorden och är det viktigaste enzymet i syntesen av allt organiskt material på planeten. Rubisco är dock ett dåligt enzym och hämmas lätt av sidoprodukter från dess katalytiska reaktioner eller av föreningar som syntetiseras av vissa växter under svaga ljusförhållanden. Rca fungerar för att lindra eller ”bota” Rubisco från sådana problematiska hämmningar. Författarna diskuterar strukturen hos Rca från olika arter och de potentiella mekanismerna för dess funktion.
Dr Mueller-Cajar ger en översikt över ”The Diverse AAA+ Machines that Repair Inhibited Rubisco Active Sites” (Mueller-Cajar). Han diskuterar förekomsten av tre evolutionärt skilda klasser av Rubiscoaktivaser (Rcas): (1) Rcas av grön och röd typ som huvudsakligen finns i fotosyntetiska eukaryoter av den gröna respektive röda plastidlinjen och (3) CbbQO som finns i kemoautotrofa bakterier. Han diskuterar utvecklingen av dessa aktivaser och deras potentiella användning i syntetisk biologi för att öka Rubiscoaktiviteten i växter.
Torsin
I sin perspektivartikel ”Torsin ATPases: Harnessing Dynamic Instability for Function” diskuterar Chase et al. Torsinerna, som också är fylogenetiskt besläktade med NBD2 i jäst Hsp104 (Chase et al.). Torsins är de enda AAA+ ATPaserna som är lokaliserade inne i ER och anslutet kärnhölje (Chase et al.). Intressant nog orsakar mutationer i TorsinA DYT1-dystoni, en neurologisk sjukdom hos människor (Chase et al.). Torsiner uppvisar en svag ATPasaktivitet som förstärks genom komplementering av den aktiva platsen på grund av sammontering med specifika accessoriska kofaktorer LAP1 och LULL1 (Chase et al.). Chase et al. föreslår att dynamisk sammansättning och upplösning av Torsin/kofaktorkomplex spelar en viktig roll för deras funktion i kärntrafiken och sammansättningen av kärnporerkomplex (Chase et al.).
Pontin och Reptin
I sin omfattande översikt om ”The Role of Pontin and Reptin in Cellular Physiology and Cancer Etiology” diskuterar Mao och Houry de många olika funktionerna hos de mycket bevarade Pontin- och Reptin AAA+ ATPaserna (Mao och Houry). Dessa två proteiner fungerar vanligtvis tillsammans som ett komplex men kan också fungera oberoende av varandra. Författarna lyfter fram Pontins och Reptins roll i kromatinombildningen. De diskuterar också hur Pontin och Reptin modulerar transkriptionsaktiviteterna hos flera proto-onkogener som MYC och β-catenin. Mao och Houry belyser hur Pontin och Reptin har visat sig vara nödvändiga för sammansättningen av PIKK-signalkomplex samt telomeras, mitotisk spindel, RNA-polymeras II och snoRNPs. Författarna avslutar med en översikt över aktuella insatser som syftar till att identifiera hämmare av Pontin och Reptin som ska utvecklas som nya anti-cancers.
Slutanmärkningar
Samlingen av 21 artiklar belyser sammanfattningsvis ett antal viktiga strukturella och mekanistiska aspekter av AAA+-proteiner som är involverade i proteinreparation och nedbrytning. Vi är spända på att se hur fältet kommer att fortsätta att utvecklas under den pågående cryo-EM-revolutionen (Egelman, 2016). Vi förväntar oss att cryo-EM kommer att möjliggöra en djupare förståelse av hur dessa fascinerande molekylära maskiner fungerar i olika situationer (Gates et al., 2017; Puchades et al., 2017; Ripstein et al., 2017; Zehr et al, 2017).
Författarbidrag
Alla angivna författare har gjort ett väsentligt, direkt och intellektuellt bidrag till arbetet och godkänt det för publicering.
Intressekonfliktförklaring
Författarna förklarar att forskningen utfördes i avsaknad av kommersiella eller finansiella relationer som skulle kunna tolkas som en potentiell intressekonflikt.
Acknowledgments
Arbetet i WH:s laboratorium inom detta område finansieras av ett projektbidrag från Canadian Institutes of Health Research (PJT-148564). JS stöds av NIH-bidrag R01GM099836.
Egelman, E. H. (2016). Den pågående revolutionen inom Cryo-EM. Biophys. J. 110, 1008-1012. doi: 10.1016/j.bpj.2016.02.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Erzberger, J. P., and Berger, J. M. (2006). Evolutionära relationer och strukturella mekanismer hos AAA+-proteiner. Annu. Rev. Biophys. Biomol. Struct. 35, 93-114. doi: 10.1146/annurev.biophys.35.040405.101933
PubMed Abstract | CrossRef Full Text | Google Scholar
Finn, R. D., Attwood, T. K., Babbitt, P. C., Bateman, A., Bork, P., Bridge, A. J., et al. (2017). InterPro 2017 – bortom annoteringar av proteinfamiljer och domäner. Nucleic Acids Res. 45, D190-D199. doi: 10.1093/nar/gkw1107
PubMed Abstract | CrossRef Full Text | Google Scholar
Gates, S. N., Yokom, A. L., Lin, J., Jackrel, M. E., Rizo, A. N., Kendsersky, N. M., et al. (2017). Ratchet-liknande polypeptidtranslokationsmekanism för AAA+ disaggregaset Hsp104. Science 357, 273-279. doi: 10.1126/science.aan1052
PubMed Abstract | CrossRef Full Text | Google Scholar
Hanson, P. I., and Whiteheart, S. W. (2005). AAA+-proteiner: har motor, kommer att fungera. Nat. Rev. Mol.Cell Biol. 6, 519-529. doi: 10.1038/nrm1684
PubMed Abstract | CrossRef Full Text | Google Scholar
Olivares, A. O., Baker, T. A., and Sauer, R. T. (2016). Mekanistiska insikter i bakteriella AAA+ proteaser och proteinomformningsmaskiner. Nat. Rev. Microbiol. 14, 33-44. doi: 10.1038/nrmicro.2015.4
PubMed Abstract | CrossRef Full Text | Google Scholar
Puchades, C., Rampello, A. J., Shin, M., Giuliano, C. J., Wiseman, R. L., Glynn, S. E., et al. (2017). Strukturen av mitokondriernas inre membran AAA+ proteas YME1 ger insikt i substratbearbetning. Science 358:eaao0464. doi: 10.1126/science.aao0464
PubMed Abstract | CrossRef Full Text | Google Scholar
Ripstein, Z. A., Huang, R., Augustyniak, R., Kay, L. E. och Rubinstein, J. L. (2017). Struktur av ett AAA+ unfoldase i processen att veckla ut substrat. Elife 6:e25754. doi: 10.7554/eLife.25754
PubMed Abstract | CrossRef Full Text | Google Scholar
Snider, J., Thibault, G. och Houry, W. A. (2008). AAA+ superfamiljen av funktionellt olika proteiner. Genome Biol. 9:216. doi: 10.1186/gb-2008-9-4-216
PubMed Abstract | CrossRef Full Text | Google Scholar
Zehr, E., Szyk, A., Piszczek, G., Szczesna, E., Zuo, X. och Roll-Mecak, A. (2017). Katanins spiral- och ringstrukturer kastar ljus över kraftslaget för mikrotubuliavskiljning. Nat. Struct. Mol. Biol. 24, 717-725. doi: 10.1038/nsmb.3448
PubMed Abstract | CrossRef Full Text | Google Scholar