Polymorfism av enstaka nukleotider
Polymorfism av enstaka nukleotider (SNP) är skillnaden mellan enstaka nukleotider i samma position i den genomiska DNA-sekvensen. I allmänhet kallas en enskild nukleotidvariation med en frekvens på mer än 1 % för en SNP. SNP:erna omfattar endast en enda basvariation, som kan orsakas av en enda basövergång eller transversion. Det finns ungefär en SNP per 1000 baser i det mänskliga genomet, och det totala antalet SNP i det mänskliga genomet är cirka 3 x 106.
SNP har blivit den tredje generationen av genetiska markörer. SNP:s egenskaper gör dem mer lämpade än andra polymorfa markörer för studier av genetisk pleiotropi i komplexa egenskaper och sjukdomar och för populationsbaserad genigenkänning. SNP har stor betydelse för biomedicinsk forskning. SNP har använts i genomövergripande associationsstudier som högupplösta markörer vid kartläggning av gener i samband med sjukdomar eller normala egenskaper. SNPs utan observerbar inverkan på fenotypen (så kallade tysta mutationer) kan också vara användbara på grund av deras kvantitet och stabila nedärvning över generationer.
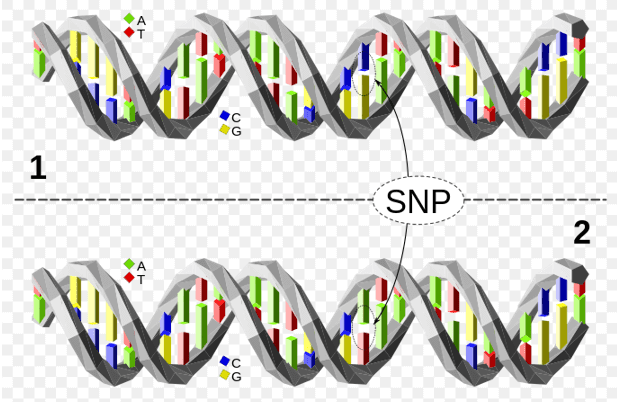 Figur 1. Den övre DNA-molekylen skiljer sig från den nedre DNA-molekylen på ett enda baspars plats (en C/A-polymorfism).
Figur 1. Den övre DNA-molekylen skiljer sig från den nedre DNA-molekylen på ett enda baspars plats (en C/A-polymorfism).
Mikroarray
DNA-mikroarray hänvisar till ett genchip med ett stort antal DNA-sekvenser för sonder i ett specifikt arrangemang som är immobiliserade på ett fast substrat. Principen är nukleinsyrahybridiseringsteorin och det detekterade provets DNA hybridiseras med DNA-mikroarrayet och förlängs. Därefter tvättas fragmenten från den icke-komplementära bindningsreaktionen på chipet bort, och genchipet utsätts för laserkonfokal skanning. Fluorescenssignalernas intensitet mäts sedan och tolkas som förekomsten av olika gener genom viss databehandling.
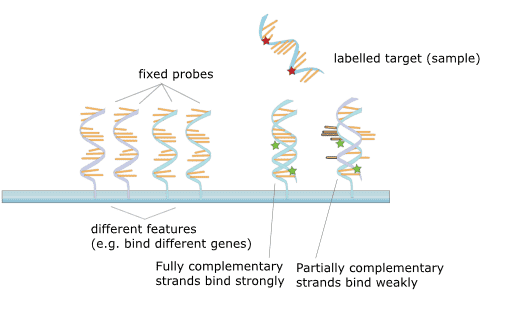 Figur 2. Principen för mikroarray.
Figur 2. Principen för mikroarray.
SNP-mikroarray
SNP-mikroarray använder kända nukleotidsekvenser som prober för att hybridisera med de testade DNA-sekvenserna, vilket möjliggör kvalitativ och kvantitativ SNP-analys genom signaldetektion. Jämfört med den traditionella diagnostiska metoden med enskilda celler är SNP-mikroarray en metod med hög genomströmning, som kan utföra tusentals reaktioner på oligonukleotidchipets yta samtidigt.
Arbetsflödet för SNP-mikroarray
Det allmänna arbetsflödet för SNP-mikroarray visas i figur 3. Kortfattat kan man säga att det finns flera processer: I korthet handlar det om följande processer: tillverkning av SNP-chip, beredning av genomiskt DNA, hybridisering och fluorescensskanning.
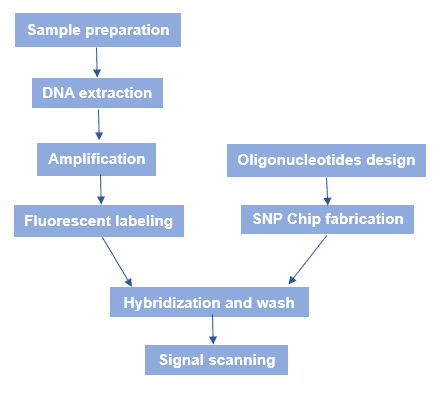 Figur 3. Arbetsflödet för SNP-mikroarray.
Figur 3. Arbetsflödet för SNP-mikroarray.
- Tillverkning av SNP-chip
De förkonstruerade oligonukleotid- eller cDNA-chipen arrangeras ordnat och med hög densitet på glasbäraren för att göra mikroarrayer eller kärnchip. Tillverkningsmetoderna för chipen omfattar ljusstyrd in-situ-syntes, kemisk spraymetod, kontaktpunktsbeläggningsmetod, in-situ-dna-kontrollerad syntes, kontaktlös mikromekanisk utskriftsmetod TOPSPOT® och mjuklitografisk reproduktion osv. Totalt 4 x 105 olika DNA-molekyler kan nu placeras på ett chip på 1 cm2. Affymetrix GeneChip®-tekniken möjliggör in situ-oligonukleotidsyntes med hög densitet och har varit en pionjär på detta område. - Förberedelse av genomiskt DNA från provet
För det första förbereds provmaterialet. Därefter extraheras det genomiska DNA:t och amplifieras genom PCR. Slutligen används olika fluorescerande färgämnen för att märka det. - Hybridisering och skanning
Det genomiska DNA som märkts med fluorescens hybridiseras med SNP-mikroarray under lämpliga reaktionsförhållanden. Efter att ha tvättats används en specifik fluorescensskanner för att skanna fluorescensen. Sedan görs datorbildanalysen för att bearbeta data innan den bioinformatiska analysen av målgenen utförs.
På CD Genomics är vi dedikerade till att tillhandahålla tillförlitliga SNP-genotyperingstjänster, inklusive genotypering genom sekvensering, SNP-mikroarray, MassARRAY SNP-genotypering, Hi-SNPseq, SNaPshot Multiplex System för SNP-genotypering, och TaqMan SNP-genotypering.
- Iwamoto K, Bundo M, Ueda J, et al. Detektion av kromosomala strukturella förändringar i enskilda celler med hjälp av SNP-arrayer: en systematisk undersökning av förstärkningsbias och optimerat arbetsflöde. Plos One, 2007, 2(12): e1306.
- Ho C C C, Mun K S, Naidu R. SNP array technology: an array of hope in breast cancer research. Malaysian Journal of Pathology, 2013, 35(1):33-43.
- Ahmad A, Iqbal M A. Significance of genome-wide analysis of copy number alterations and UPD in myelodysplastic syndromes using combined CGH-SNP arrays. Current Medicinal Chemistry, 2012, 19(22).
- Sund K L, Zimmerman S L, Thomas C, et al. Regions of homozygosity identified by SNP microarray analysis aid in the diagnosis of autosomal recessive disease and incidentally detect parental blood relationships. Genetics in Medicine Official Journal of the American College of Medical Genetics, 2013, 15(1):70-78.
- Liu S, Zhou Z, Lu J, et al. Generation of genome-scale gene-associated SNPs in catfish for the construction of a high-density SNP array. Bmc Genomics, 2011, 12(1):53.